RNA干扰作用原理及其应用
【关键词】 RNA干扰 siRNA dsRNA RNA诱导的沉默复合体 Argonaute RNA聚合酶III启动子
【Abstract】 RNA interference(RNAi) in diverse organisms reveals the same highly conserved mechanism with an ancient origin.RNAi is the mode of sequence-specific post-transcriptional gene silencing in animals and plants initiated by homologous double-stranded RNA(dsRNA).The discovery of RNAi and the molecular mechanism will help us apply it to study the gene function and exploit the gene drug.
【Key words】 RNA interference(RNAi) small interfering RNA(siRNA) double-stranded RNA(dsRNA) RNA-induced silencing complex(RISC) Argonaute pol III
1 背景
20 多年前,在对矮牵牛(petunias)进行的研究中有个发现:Rich Jorgensen和同事将一个能产生色素的基因置于一个强启动子后,导入矮牵牛中,试图加深花朵的紫颜色,结果没看到期待的深紫色花朵,多数花成了花斑的甚至白的。因为导入的基因和其相似的内源基因同时都被抑制,Jorgensen将这种现象命名为协同抑制(cosuppression)。在真菌中也有类似的现象,1996年就在脉孢菌属(Neurospora)发现这种现象。当时将这种现象命名为基因表答的阻抑作用(quelling)[1]。
通过对转基因植物的研究,发现相应基因的转录并不受影响,将这种现象称为基因转录后沉默(posttranscriptional gene silencing, PTGS)。转录后的基因沉默可能是进化过程中一种抵御转座子和RNA病毒的防御机制,可能在植物和动物分化之前就已经出现。1998年华盛顿卡耐基研究院的Andrew Fire 和马萨诸塞大学癌症中心的Craig Mello 首次将双链dsRNA (double-stranded RNA)注入线虫,结果诱发了比正义链和反义链的单独注射都要强的基因沉默。将这种由dsRNA(double-stranded RNA)引发的特定基因表达受抑制现象称为RNA干扰作用(RNA interference RNAi)[2]。
2 RNAi的分子机制
dsRNA(double-stranded RNA)可以介导基因沉默作用,以dsRNA为基点研究基因沉默的分子机制成为热点。dsRNA指的是大于30个碱基对的RNA分子。哺乳动物细胞有至少2条路径竞争双链RNA(dsRNA),其一是特异性路径:特殊dsRNA的序列用于RNAi,起始阶段dsRNA被切成siRNA(small interfering RNA 或short interfering RNAs)。siRNA是RNA干扰作用赖以发生的重要中间效应分子,能提供一定的信息,允许一个特定的mRNA被降解。siRNA正义链与反义链各有21个碱基,其中19个碱基配对,再每条链的3’端都有2个不配对的碱基(图1)。

图1 siRNA
另一条是非特异性路径:只要有长的dsRNA的存在它可以降解所有的RNA,抑制所有蛋白质的合成。长的dsRNA激活蛋白激酶PKR,激活的PKR通过一系列的磷酸化关闭翻译起始因子,导致翻译抑制。也可以通过激活2’-5’AS 合成一个分子激活RNase L,导致非特异的RNA降解[3]。
关于特异性的RNA作用机制模型,包括起始阶段和效应阶段[4]。起始阶段dsRNA在Dicer酶(RNaseIII家族中特异识别双链RNA的一员,属内切核酸酶)的作用下加工裂解21-23核甘酸长的小分子干扰RNA片断(siRNA)[5]。Dicer含有解旋酶活性以及dsRNA结合域和PAZ结构。
在RNAi 的效应阶段,siRNA双链结构解旋并形成有活性的蛋白/RNA复合物(RNA-induced silencing complex or RISC),在siRNA 解双链即RISC激活过程需一个ATP[5]。由RISC中siRNA反义链与mRNA互补区域结合,随后切割mRNA从而达到在RNA水平干扰基因表达。RISC由多种蛋白成分组成,包括核酸酶,解旋酶和同源RNA链搜索活性等。
最新研究表明,Argonaute蛋白为RISC的特有成分,被比喻成“切薄片的机器”。Argonaute有一个月牙型的底座由三部分组成,分别为N-端(amino-terminal)、中部(middle)和PIWI区域(PIWI domain)。在月牙底座上面有一个PAZ部分,由茎杆结构支撑。Argonature蛋白有一个明显的凹槽贯穿整个蛋白,凹槽内壁带正电有利于与RNA带负电的磷酸骨架结合。siRNA解旋后, siRNA单链的3’端伸入PAZ的裂缝中,整个单链沿着PAZ伸展开,同时目的片段mRNA结合于新月型底座。mRNA与siRNA互相配对形成双链后,在离单链siRNA5’端9个碱基处(即离siRNA3’端11-12个碱基处)切割mRNA(图2)[6]。
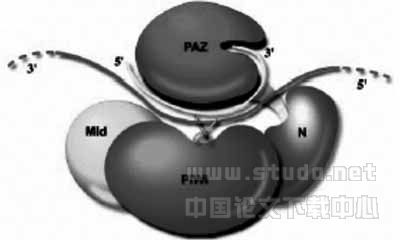
图2 siRNA引导mRNA被切割的模型
3 siRNA的表达
最便利的siRNA表达载体用三种RNA聚合酶III 启动子(pol III)中的一种(Sui 2002 ,Lee 2002,Paul 2002),操纵一段小的发夹RNA(short hairpin RNA, shRNA)在哺乳动物细胞中的表达(图3)[7]。
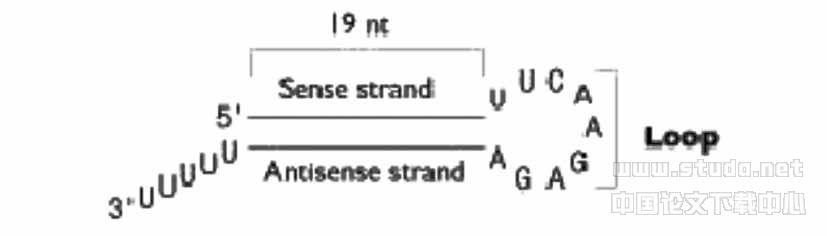
图3 hairpin siRNA
这类启动子有大家熟悉的人源和鼠源的U6启动子和人H1启动子[8]。采用RNA pol III启动子是因为它结构简单,而且可在哺乳动物细胞中表达小分子RNA,并且它可通过一串(3-6个)U来终止转录的,这刚好符合原始设计的siRNA的3’端含有2个突出碱基U( Elbashir 2002)。使用这类载体,首先要设计2段编码短发夹RNA序列的DNA单链,然后订购这2段DNA单链,退火,克隆到相应载体的pol III 启动子下游。克隆可能要几周甚至数月的时间,为保证克隆的序列的正确性,还要进行测序。
病毒载体用于siRNA表达,其优势在于可以直接高效率感染细胞并维持较长时间的基因沉默[9]。
siRNA目标位点的筛选是实现RNAi作用的关键,一般有以下几个原则[10,11]:(1) 在预定沉默的mRNA中找一段21nt的序列,起始是AA;(2)找2~4个小片段的RNA,通过SECs(siRNA expression cassettes,SECs)筛选最有效的siRNA;(3)在19nt的RNA片断中不能含有连续4个T或A的区域;(4)通过BLAST 确定片断的特异性;(5)片断GC%应该在30%~50%。4 RNAi的应用前景
4.1 研究信号传导通路和基因功能 RNAi能够在哺乳动物中降低特异性基因的表达,制作多种表型,而且抑制基因表达的时间,控制基因表达的部位。
4.2 开展基因的新途径 一基因家族的多个基因具有一段同源性很高的保守序列这一特性, 设计针对这一区段序列的dsRNA分子,只注射一种dsRNA即可以产生多个基因表达同时降低的表现,也可同时注射多种dsRNA而将多个序列不相关的基因同时剔除。为治疗多基因调控的肿瘤疾病,带来新的治疗策略。
【】
1 Isolation of quelling-defective (qde) mutants impaired in posttranscriptional transgene-induced gene silencing in Neurospora crassaProc. Natl Acad Sci USA,1997,94:10233-10238.
2 Fire A ,Xu S,Montgomery M K,et al.Potent and specific genetic interferenceby double-strand RNA in coenorhabditis elegans.Nature,1998,391(6669):744-745.
3 Nature,2001,411,428-429.
4 Matzke.M, Matzke A J, Kooter J M.RNA:guiding gene silencing. Science,2001,293(5532):1080-1083.
5 Nykanen A,Haley B,Zamore P D.ATP requirements and small interfering RNA structure in the RNA interference pathway .Cell,2001,107(3):309-3021.
6 Ji-Joon Song, Stephanie K. Smith Gregory J. Hannon,1 Leemor Joshua-Tor1,2*3 Crystal Structure of Argonaute and Its Implications for RISC Slicer Activity SCIENCE,2004,305.
7 Yu J.-Y.,DeRuiter S.L.,Turner,D.L. RNA interference by expression of short-interfering RNAs and hairpin RNAs in mammalian cells. Proc Natl Acad Sci USA,2002,99(9) : 6047-6052.
8 Miyagishi M, Taira K. U6 promoter-driven siRNAs with four uridine 3’ overhangs effectively suppress targeted gene expression in mammalian cells. Nature Biotechnology,2002,20 : 497-500.
9 Gustavo Tiscornia, Oded Singer,Masahito Ikawa, et al. A general method for gene knockdown in mice by using lentiviral vectors expressing small interfering RNA Proc Natl Acad Sci USA,2003,100(4): 1844-1848.
10 Brown D, Jarvis R, Pallotta V, et al. RNA interference in mammalian cell culture: design, execution, and analysis of the siRNA effect. Ambion TechNotes 9(1): 3-5.
11 Sui G,Soohoo C,Affar EB, et al.A DNA vector-based RNAi technology to suppress gene expression in mammalian cells. Proc Natl Acad Sci. US A,2002,99(8): 5515-5520.