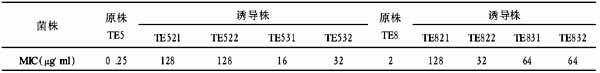
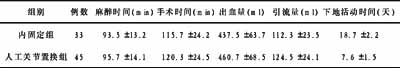
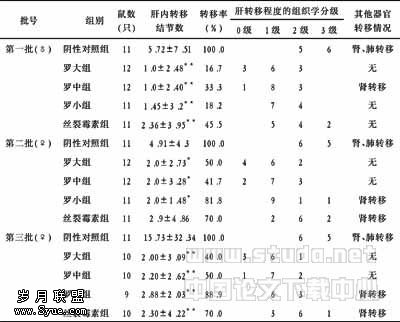
幽门螺杆菌Ⅳ型分泌系统cagX基因的克隆及生物信息学特征预测
【摘要】 目的: 构建表达幽门螺杆菌(Helicobacter pylori,H. pylori) NCTC 11637 IV型分泌系统cagX(HP0528)全长编码基因的原核表达载体。方法: 用PCR方法从H. pylori基因组DNA中扩增cagX编码基因片段,将cagX克隆至pGEM-T载体,转化大肠杆菌DH5α,再将其定向插入pET32a(+)载体中,经IPTG诱导表达。SDS-PAGE分析表达结果,并进行序列测定和生物信息学预测。结果: 克隆幽门螺杆菌NCTC 11637 cagX 基因全长1 569 bp(基因库登录号为EF608160),编码522个氨基酸,融合蛋白的相对分子质量约为80.5 kD。与基因库公布的其他H. pylori菌株基因序列的核苷酸同源性为96%~99%。预测结果显示H. pylori 11637,26695,J99间二级结构有一定差异,但抗原性相同。结论: 成功克隆并表达了H. pylori NCTC 11637 cagX 基因,并对其生物学特征进行了预测,为进一步研究其生物学功能奠定了基础。
【关键词】 幽门螺杆菌; cagX; IV 型分泌系统; 生物信息; 序列分析
[Abstract] Objective: The prokaryotic expression vector for gene encoding the fusion protein cagX of type IV secretion system(TFSS) was constructed and expressed. Methods: H. pylori cagX gene was amplified by PCR using the genome DNA. The PCR product was inserted into pGEM-T vector and then transformed into E.coli DH5α. The positive recombinant clone was analyzed by digestion of restriction endonuclease. Then the cagX gene fragment was inserted directionally into vector pET32a(+) to construct recombinant clones of cagX.After they were expressed by BL-21 E.coli cells, the PCR products were sequenced and analyzed. Results: A 1569 base pairs long cagX gene, which encodes a product of 522 amino acid, was obtained using PCR method and was cloned into pGEM-T vector successfully. It was found that the relative molecular weight of this fusion protein was approximately about 80.5 kD. GenBank accession number is EF608160.The sequence analysis for cagX showed that it shares 96%~99% homology with other strains in GenBank. The result of analysis revesled that there were certain differences in secondary structure, but the antigenicity was identical. Conclusion: It was indicated that we had obtained the correct cagX gene, which posed a basis for further researching on its biological function.
[Key words] Helicobacter pylori; cagX; type IV secretion system;bioinformatics;sequence analysis
幽门螺杆菌(Helicobacter pylori, H. pylori)是一种定植于人类胃粘膜的螺旋状、革兰阴性微需氧菌,现已被公认为慢性活动性胃炎和消化性溃疡的重要病因,并与肠型胃癌及胃MALT淋巴瘤的发生密切相关[1-3]。I型H. pylori菌株中含有一个约40 kb的cag致病岛(cytotoxin associated gene pathogenicity island,cag PAI)。cag致病岛基因呈高密度分布并编码一个分泌转运系统(type IV secretion system,TFSS)。根癌农杆菌(Agrobacterium tumefaciens,A.tumefaciens)的VirB-VirD系统或称Ti-DNA输送系统被公认为是TFSS的标准,它是由VirB操纵子的产物(VirB1-VirB11蛋白)和VirD4基因编码的产物VirD4装配而成,形成跨膜通道将蛋白直接注入宿主细胞内[4]。H. pylori中VirB9的同系物cagX/HP0528由cag致病岛编码,Tanaka等[5]用免疫金标记扫描电镜技术发现,VirB9的同系物HP0528/CagX定位于H. pylori的外膜上,沿着菌毛的长度定植,并与HP0532/CagT通过分子间二硫键交联形成外膜复合物。
本文首次报道了H. pylori NCTC 11637 cagX 基因全序列,并对cagX序列进行了生物信息学比较分析,为进一步研究cagX基因的生物学功能奠定基础。
1 材料和方法
1.1 材料
1.1.1 菌株及质粒 H. pylori NCTC 11637购自预防院流行病学研究所;大肠埃希菌DH5α,BL21,pET32a(+)载体由江苏大学医学技术学院中心实验室保存;pGEM-T载体购自Promega公司。
1.1.2 试剂和仪器 哥伦比亚培养基、厌氧袋(Oxoid公司);Ex-Taq DNA聚合酶,dNTPs,T4DNA连接酶(快速连接试剂盒)TakaRa Agarose Gel DNA Purification Kit Ver 2.0,DL 2000 DNA标准参照物,λ-EcoT14 I digest DNA标准参照物(TakaRa公司);限制性核酸内切酶EcoR I及Sal I(Fermantas公司);DL 3000 DNA标准参照物(上海伯叟生物科技有限公司);1 kb DNA Ladder(Toyobo);PCR基因扩增仪(Mastercycler Gracent, Eppendor公司)、自动凝胶成像系统(美国UVP公司),其他常规试剂按照《分子克隆实验指南》要求配制。
1.2 方法
1.2.1 H. pylori基因组DNA提取 H. pylori NCTC 11637 标准菌株按常规微需氧方法培养。刮取96 h培养的菌落,按以下步骤提取DNA:用1 ml PBS收集细菌,并重悬细胞,13 000 r/min离心5 min; 100 μl TE悬浮细胞,加入500 μl GES混匀,13 000 r/min离心5 min;将上清移至洁净管中,加入650 μl氯仿混匀,离心5 min;将上清移至洁净管中,加入0.54倍体积的异丙醇,混匀,室温5 min;离心1 min,弃上清,用500 μl 70%乙醇洗涤,干燥后用100 μl TE重悬。于-20℃保存备用。
1.2.2 引物合成 根据基因库中已报道的H. pylori 26695 NC_000915的cagX基因(HP0528基因)全序列,利用软件Primier 5. 0设计PCR引物:上游引物P1:5′-GGAATTCATGGGGCAGGCATTTTTTA-3′(26 bp);下游引物P2:5′-GGGTCGACTTATTTATCTCTGACAAGAGGGAG-3′(32 bp)。
在上游引物5′端加EcoR I酶切位点;在下游引物5′端加Sal I酶切位点。预期的PCR产物长度为1 569bp。引物由上海生工生物技术有限公司合成。
1.2.3 PCR扩增及纯化 以H. pylori NCTC 11637基因组DNA为模板,P1和P2为引物,用Ex-Taq聚合酶进行PCR扩增(在50 μl反应体系中,分别加入dNTP 4 μl,Mg2+3 μl,10×缓冲液(Mg2+阴性)5 μl,DNA模板2 μl,浓度为20 μmol/L的P1和P2引物各1.4 μl,Taq聚合酶0.4 μl,ddH2O 33.4 μl,按照PCR条件扩增),扩增条件为:94℃预变性5 min后,按94℃ 45 s,64℃ 45 s,72℃ 2 min,35个循环,最后72℃延伸10 min。PCR产物经1.0%琼脂糖凝胶电泳,EB染色,Genius凝胶电泳图像分析系统分析鉴定。
1.2.4 TA克隆及鉴定 胶回收试剂盒回收目的片段,胶回收PCR产物与pGEM-T载体(摩尔比8∶1),加1 μl T4DNA连接酶,4℃连接过夜。连接产物转化DH5α感受态细胞。转化后的细菌涂布于含50 μg/ml 氨苄青霉素的LB平板上,37℃培养16 h。挑取菌落转种于含氨苄青霉素50 μg/ml的LB液体培养基,37℃ 振摇12 h,用碱裂解法进行质粒小量提取,并进行EcoR I,Sal I双酶切鉴定和质粒PCR鉴定。
1.2.5 重组质粒的构建 将质粒pET32a(+)和TA克隆鉴定阳性的质粒经EcoR I和Sal I双酶切,回收酶切片段,T4DNA连接酶试剂盒作用下16℃连接过夜,转化宿主菌BL21,双酶切鉴定筛选出阳性克隆。
1.2.6 目的蛋白的表达 取经过酶切鉴定阳性的重组工程菌接种到含氨苄青霉素的LB液体培养基中,37℃培养至D(600 mm)达到0.6~0.8时,加入终浓度为1 mmol/L IPTG诱导4 h。采用SDS-PAGE分析pET32a-CagX融合蛋白的表达,用凝胶成像系统分析重组蛋白的相对含量。
1.2.7 DNA序列测定与生物信息学分析 将经过鉴定的阳性重组质粒送至上海生工测序。测序结果通过基因库进行同源性比较分析。用DNA Star 5.0软件对cagX基因核苷酸序列及其推导的氨基酸序列与基因库中的不同菌株的幽门螺杆菌cagX基因进行比较,分析它们之间的同源性及变异程度。
使用DNA Star软件中的Protean程序H. pylori NCTC 11637 CagX蛋白的分子质量、氨基酸组成,预测其等电点。同时预测H. pylori J99和H. pylori 26695的二级结构和抗原性,以探讨cagX基因在不同菌株间的变异性。
2 结果
2.1 cagX基因PCR扩增结果
PCR结果发现在1 569 bp左右有一条带,与预计大小一致,无非特异性条带,如图1所示。
1:DNA 标准(DL 3 000); 2:cagX PCR产物;3.阴性对照
图1 cagX基因PCR产物(略)
Fig 1 cagX gene PCR product by agarose gelelectrophoresis
2.2 TA克隆重组质粒的筛选与鉴定
将PCR产物与pGEM-T载体连接产物转化至DH5α,随机挑取菌落,抽提质粒,双酶切产物电泳后,阳性克隆出现的两条带分别位于3 003 bp和1 569 bp处,如图2所示。
2.3 重组表达质粒的酶切鉴定
重组质粒pET32a(+)-cagX经EcoR I及Sal I双酶切鉴定结果,如图3所示。
2.4 目的蛋白的表达
向培养至D(600 mm)达到0.6~0.8的阳性重组工程菌液中,加入IPTG诱导,其全菌蛋白的SDS-PAGE鉴定结果,在相对分子质量约80.5 kD处可见明显的外源蛋白表达带,与预计相符,如图4所示。
1:DNA 标准(DL 2 000);2:PCR产物;3:pGEM-T/cagX/EcoR I+Sal I;4: pGEM-T/cagX/EcoR I; 5:λ-EcoT14 I digest DNA 标准
图2 重组质粒pGEM-T /cagX 的双酶切鉴定结果(略)
Fig 2 Double restriction enzyme digestion map of the recombinant plasmid pGEM-T /cagX
1:DNA 标准(DL 3 000) ;2:cagX PCR产物;3:pET32a(+) /cagX/EcoR I+Sal I;4:pET32a(+) /cagX/EcoR I;5:pET32a(+) /cagX/Sal I;6:1 kb DNA Ladder
图3 重组质粒pET32a(+) /cagX 的双酶切鉴定结果(略)
Fig 3 Double restriction enzyme digestion map of the recombinant plasmid pET32a(+) /cagX
2.5 cagX基因的测序结果及编码蛋白分子特征
将经鉴定后的重组质粒送往上海生工进行核苷酸序列测定,DNA测序结果表明,构建的重组质粒上的cagX长度为1 569 bp,GC含量为35.05%,编码522个氨基酸,基因库登录号为EF608160。推导出的蛋白分子质量为60.5 kD,含有92个强碱性氨基酸(K,R),71个强酸性氨基酸(D,E),167个疏水性氨基酸(A,I,L,F,W,V),143个极性氨基酸(N,C,Q,S,T,Y),等电点为9.467。
1.IPTG诱导前;2.IPTG诱导4 h后;3.蛋白质标准
图4 融合蛋白pET32a(+)-CagX的诱导前后全菌SDS-PSGE图(略)
Fig 4 SDS-PAGE analysis of pET32a(+)-CagX fusion protein
2.6 H. pylori NCTC 11637 cagX基因的生物信息分析
2.6.1 cagX基因核酸序列和氨基酸序列分析 将测序结果通过基因库与已知的两种国际标准菌株H. pylori 26695和J99的HP0528基因的核苷酸序列进行比对。与26695 菌株的HP0528比较,同源性为98%(1538/1569),碱基总数相同;与J99 菌株的cagX比较,同源性为97%(1527/1571)。根据3种菌株的氨基酸序列,应用MagAlign程序构建的氨基酸序列差异表显示各菌株间氨基酸序列同源性为98.3%~98.7%(图5)。
图5 3株幽门螺杆菌的氨基酸序列同源性(上)和变异性(下)(略)
Fig 5 Amino acid sequence homology and divergence of 3 H. pylori Strains
2.6.2 3种菌株的CagX蛋白二级结构预测分析 用Chou-Fasman法分析H. pylori 26695,J99和NCTC 11637的二级结构,如图6所示。发现:3株的α螺旋相同;β折叠的差异表现在NCTC 11637在18~21位比其他2株少了1个β折叠区域,而H.phlori26695在251~255位比其他2株少了1个β折叠区域;3株的β转角基本相同,其差异性表现在NCTC 11637 在20~23位比其他俩株多含有1个β转角,而461~464位比其他俩株少含有1个β转角。
2.6.3 3种菌株的CagX蛋白抗原性预测分析 本研究运用Jameson-Wolf法对3种菌株的抗原性进行了分析,如图7所示。结果显示重组蛋白有多个明显的具有抗原活性的结构域,且3种菌株间的抗原性无差异。
图6 Hp 11637/Hp 26695/Hp J99 CagX的二级结构预测(略)
Fig 6 Prediction of secondary structure of CagX protein of Hp 11637/Hp 26695 and Hp J99
图7 Hp 11637/Hp 26695/Hp J99 CagX的抗原性预测(略)
Fig 7 Antigenic prediction of CagX protein of Hp 11637/Hp 22695 and Hp J99
3 讨论
根癌农杆菌的VirB-VirD系统或称Ti-DNA输送系统被公认为是TFSS的标准,它是由virB操纵子的产物(VirB1-VirB11蛋白)和virD4基因编码的产物VirD4装配而成,形成跨膜通道将蛋白直接注入宿主细胞内[4]。Tanaka等[5]认为H.pylori的TFSS装置是伸出细菌外膜表面的丝状大分子结构。在H. pylori中,TFSS组分包括定位于内膜的ATPases(HP0544/CagE和HP0525/Cagα,以及偶联蛋白HP0524/Cagβ,它们分别是VirB4,VirB11及VirD4的同源物),为转运底物提供能量[6]。HP0529/CagW的突变表型和预测存在5或6个跨膜螺旋,推测其功能可能类似于VirB6。HP0523可能与VirB1的功能相似,可能是一个糖基转移酶,切断肽聚糖,使得TFSS装置能够横跨细菌细胞壁。跨膜通道本身连接内外膜,由HP0529/VirB6,HP0532(CagT)/VirB7, HP0530/VirB8,HP0528(CagX)/VirB9和HP0527/VirB10组成[7]。Tanaka等[5]用免疫金标记扫描电镜技术发现,VirB7和VirB9的同系物HP0532/CagT和HP0528/CagX沿着菌毛的长度定植。在根癌农杆菌中,VirB7自身通过cys-24形成二硫键交联的同型二聚体,VirB9的cys-262与VirB7的cys-24间形成二硫键交联的异型二聚体,然后与内膜蛋白VirB8和VirB10作用形成一个稳定的跨越内膜和肽聚糖层而直达外膜内表面的通道[8,9]。
目前H. pylori中由cag致病岛编码的蛋白构成的IV型分泌系统已逐渐成为公认的H. pylori重要的毒力因子。通过转运细胞相关毒素cagA进入胃上皮细胞、诱导促炎细胞因子IL-8的表达,在H. pylori致病中起着关键作用[10,11]。
本文从H. pylori NCTC11637 菌株中克隆cagX全长编码基因并对其DNA序列进行测定,利用生物信息数据库及生物学软件对cagX基因序列进行同源性分析。结果表明,cagX基因全长1 569 bp,编码522个氨基酸,融合蛋白的相对分子质量约为80.5 kD;是一类高度保守的原核型基因,与H. pylori其他菌株的同源性在96%~99 %,与真核生物无同源序列。3种标准菌株间氨基酸序列的差异导致的二级结构有很小的差异,但3种菌株的抗原性完全相 同。本研究表明cagX基因成功克隆并表达,为进一步研究cagX基因的生物学功能及潜在的临床应用前景等奠定了实验基础。
【】
[1] Cover TL, Blaser MJ. Helicobacter pylori and gastroentoenal disease[J]. Annu Rev Med, 1992, 43: 135-145.
[2] Correa P. Human gastric carcinogenesis: a multistep and multifactorial process-first american cancer society award leature on cancer epidemiology and prevention[J]. Cancer Res, 1992, 52(24): 6735-6740.
[3] Sanders MK, Peura DA. Helicobacter pylori-associated diseases[J]. Curr Gastroenterol Rep, 2002, 4(6):448-454.
[4] Cascales E, Christie PJ. The versatile bacterial type IV secretion systems[J]. Nat Rev Microbiol, 2003, 1(2):137-149.
[5] Tanaka J, Suzuki T, Mimuro H, et al. Structural definition on the surface of Helibobacter pylori type IV secretion apparatus[J]. Cell Microbiol, 2003, 5(6):395-404.
[6] Yuan Q, Carle A, Gao C, et al. Identification of the VirB4-VirB8-VirB5-VirB2 pilus assembly sequence of type IV secrection systems[J]. J Biol Chem, 2005, 280(28): 26349-26359.
[7] Busler VJ, Torres VJ, McClain MS, et al. Protein-protein interactions among Helicobacter pylori cag proteins[J]. J Bacteriol, 2006, 188(13): 4787-4800.
[8] Das A, Xie YH. The Agrobacterium T-DNA transport pore proteins VirB8,VirB9, and VirB10 interact with one another[J].J Bacteriol, 2000, 182(3):758-763.
[9] Anderson LB,Hertzel AV,Das A. Agrobacterium tumefaciens VirB7 and VirB9 form a disulfide-linked protein complex[J]. Proc Natl Acad Sci USA, 1996,93(17): 8889-8894.
[10] Li SD, Kersulyte D, Lindley IJ, et al. Multiple genes in the left half of the cag pathogenicity island of Helicobacter pylori are required for tyrosine kinase-dependent transcription of interleukin-8 in gastric epithelial cells[J]. Infect Immun, 1999, 67(8):3893-3899.
[11] Asahi M, Azuma T,Ito S,et al.Helicobacter pylori CagA protein can be tyrosine phosphorylated in gastric epithelial cells[J]. J Exp Med,2000, 191(4): 593-602.