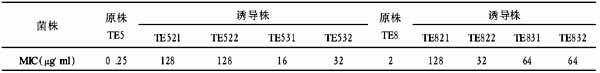
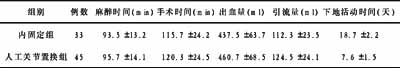
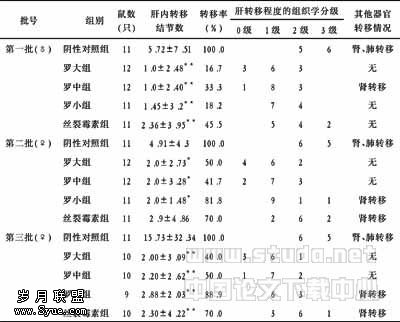
受体型酪氨酸激酶RONΔ160对NIH-3T3细胞生长和移动浸润能力的作用
【摘要】 目的研究受体型酪氨酸激酶RONΔ160对3T3细胞生长和移动浸润能力的作用。方法 NIH-3T3细胞转染质粒pDR2-RONΔ160,建立稳定表达RONΔ160的3T3-RONΔ160细胞株;免疫沉淀法检测细 胞RONΔ160的酪氨酸磷酸化;倒置显微镜观察细胞形态变化;软琼脂培养法观察RONΔ160对NIH-3T3细胞生长能力的作用;体外跨室趋化运动实验检测RONΔ160对NIH-3T3细胞移动浸润能力的作用。结果成功建立3T3-RONΔ160细胞株,western blot证明其稳定表达RONΔ160蛋白;免疫沉淀试验结果显示RONΔ160 具有自身酪氨酸磷酸化;软琼脂生长实验示转染组细胞生长克隆数为146.00±3.61,而未转染组和载体对照组分别为11.67±1.15和12.67±2.52,转染组与未转染组、载体对照组分别比较,差异均有统计学意义(t分别=51.60、151.19,P均<0.05);体外跨室趋化运动实验结果示转染组穿膜细胞数为458.50±4.95,而未转染组和载体对照组分别为20.00±5.66和21.50±4.95,转染组与未转染组、载体对照组分别比较,差异均有统计学意义(t分别=58.47、437.00,P均<0.05)。结论表达受体型酪氨酸激酶RONΔ160可使NIH-3T3细胞形态明显变化,并显著增强NIH-3T3细胞的生长能力和移动浸润能力。
【关键词】 蛋白质体酪氨酸激酶 细胞生长 肿瘤浸润
【Abstract】 Objective To investigate the roles of RONΔ160receptor tyrosine kinase in growth and motile/invasive ability ofNIH-3T3cell line.Methods NIH-3T3cell was transfectedwith plasmid pDR2-RONΔ160to construct a3T3-RONΔ160cell line stably expressing RONΔ160protein;the cell morphology change was observed with convert mi-croscopy;thetyrosine phosphorylation of RONΔ160was measured by immunoprecipitationmethod;cell growth and motile/invasive ability were tested by soft agar and migration assay.Results 3T3-RONΔ160cell line stably expressing RONΔ160proteinwas obtained successfully;transfected cells were smaller spindle-shaped compared with untransfected cells and vector control cells;soft agar results showed growth cell clones ofNIH-3T3,3T3-RONΔ160and3T3-pDR2were146.00±3.61,11.67±1.15and12.67±2.52(t=51.60,151.19,P<0.05);transwell migration assayresults showedmigrated cells ofNIH-3T3,3T3-RONΔ160and3T3-pDR2were458.50±4.95,20.00±5.66and21.50±4.95(t=58.47,437.00,P<0.05).Conclusions Expression of RONΔ160led to the morphology change and the in-censement ofgrowth and motile/invasive abilities ofNIH-3T3cells.
【Key words】protein-tyrosine kinase;cell growth;neoplasm invasiveness
受体型酪氨酸激酶(recepteurd'origine nantais,RON)属于MET原癌基因家族成员,被广泛认为在调节肿瘤细胞的运动和浸润能力中起重要作用。RON蛋白是属于Ⅰ型跨膜蛋白的大小为180kD的异源二聚体,由40kD的胞外区和含酪氨酸激酶活性的150kD的跨膜区组成 [1] 。RON在巨噬细胞刺激蛋白(macrophage-stimulating protein,MSP)刺激下酪氨酸 全科医学临床与2007年7月第5卷第4期Clinical Education of General Practice Jul.2007,Vol.5,No.4
·302·
磷酸化而活化 [2] 。RONΔ160是RON缺失外显子5、6的变异体,能够自身酪氨酸磷酸化而活化。RON及其变异体对调节细胞的生长、分化和生存有重要作用,同时也参与细胞转化、肿瘤的与转移 [3] 。本研究就RONΔ160对调节NIH-3T3细胞的生长、转化和运动浸润能力做初步研究。
1材料和方法
1.1材料
1.1.1主要试剂 包括:Lipofectamine转染试剂盒(由美国Invitrogen公司生产,批号KHVY01)、Hy-gromycin B(由美国Roche公司生产,批号11231400)、DMED培养液和胎牛血清(由美国Gibco公司生产)、MSP、兔IgG抗RONβ链C-terminal peptide和单抗ID2(鼠mAb抗RONβ链胞外段)(由王明海教授惠赠)、HRP标记的羊抗兔IgG(由美国Jackson公司生产,批号J0S1GF2)、电化学发光底物(由美国Amer-sham公司生产)、聚偏氟乙烯膜(PVDF,由美国Milli-pore公司生产)、β-actin单抗,琼脂糖(由美国sigma公司生产)、48孔标准趋化小室(Standard48Well Chemotaxis Chamber)和聚碳酸脂微孔滤膜(Polycarbonate membranes10μm pore)(由美国Neuro ProbeInc.公司生产)。
1.1.2主要仪器包括:CO 2 孵箱(HERA cell,Her-aus);倒置显微镜(MPS30,Leica)、电热恒温水浴箱(SC-15,宁波新芝)、蛋白印迹设备(Bio-Rad);蛋白印迹成像系统(VersaDoc,Bio-Rad)。
1.1.3细胞株与质粒 NIH-3T3为本实验室所购存。插入RONΔ160全长cDNA的真核表达载体pDR2(pDR2-R0NΔ160)为王明海教授惠赠。
1.2方法
1.2.1 细胞转染与克隆化培养 取对数生长期的NIH-3T3细胞(70%~80%融合)5×l0 4 /孔接种于24孔板中培养24h后,加入hygromycinB,终浓度为0、50、100、150、200、250、300、400、500、600、800、1000mg/L,观察14d,获得最小抑制生长浓度为400mg/L。取对数生长期的NIH-3T3细胞(70%~80%融合)接种于6cm板中,24h后培养至80%融合。重组质粒pDR2-R0NΔ1605μg与lipofectamineTM5μl混匀后转染细胞,6~24h更换普通培养基,培养24h,以100∶l稀释传代至10cm直径的培养皿以400mg/L Hy-gromycinB选择,培养7~10d待单个克隆肉眼可见时,以0.25%的胰酶消化挑选克隆。选择培养基中扩大培养后检测目的基因R0NΔ160的表达情况。同时以转染 空载体的细胞(3T3-pDR2)作载体对照。
1.2.2 Westen印迹收获培养细胞后,蛋白提取液(3×10 6 细胞裂解于200μl裂解缓冲液,50mmol/L Tris,pH7.45,0.15mmol/LNaCL,2mmol/LEDTA,0.5%NP-40,0.5%TritonX-100,100μmol/Lsodiumvana-date,5mg/Lleupeptin,aprotinin,and soybeantrypsin in-hibitor)抽提蛋白,4℃,15000r/min离心15min,取上清液并定量。每孔蛋白量50μg加上样缓冲液后煮沸5min上样。进行8%十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS—PAGE),转至聚偏氟乙烯膜(PVDF),封闭,先后加入I抗、Ⅱ抗孵育,显色(化学发光底物ECLA液1mL+ECLB液1mL),再用VersaDoc成像系统观察分析。
1.1.3免疫沉淀收获培养细胞后,MSP阳性组用MSP2nm刺激10min,然后与MSP阴性组一起用蛋白提取液抽提蛋白并定量;等量蛋白与ID2单抗1μg混合后再与G-Sepharose磁珠混合,4℃摇床混匀过夜,洗涤3~5次后,在含2-ME的样品缓冲液中煮沸3~5min洗脱沉淀蛋白,行SDS—PAGE跑胶并用兔IgG抗RONβ链C-terminal peptide作为I抗行western blot显色观察。
1.1.4软琼脂培养基生长试验 使用含2%FBS的DMEM培养液配制0.6%的琼脂溶液,60℃预热后,取2.5ml铺于每个6cm板底部,冷却后形成下层培养基;用含2%FBS的DMEM培养液配制0.25%的琼脂溶液,于50℃水浴预热,待冷却至40~42℃时,收集细胞并计数,5×10 3 细胞加入1ml0.25%冷却的琼脂溶液,混匀后立即铺于下层培养基上,继续冷却后形成上层培养基。置于5%CO 2 的37℃培养箱,每5~7天加入1mlDMEM培养液,2~3周后观察结果,计数每个培养板总数,表示细胞的生长能力。每种细胞分MSP2nm刺激与不刺激2组,平行实验3次。
1.1.5体外跨室趋化运动能力的测定趋化小室下室每室加入29μlDMEM无血清培养基。覆盖Ⅳ型胶原包被的聚碳酸脂微孔滤膜和乳胶隔垫,安装好上室。调整收获的细胞悬液浓度为4×10 8 cells/L,上室加入收获的细胞悬液每室45μl,每组2个复孔。37℃、5%CO 2 孵育5h,取出聚碳酸脂微孔滤膜,滤膜上面细胞用特殊刮膜器刮除干净,下层粘附的穿膜细胞以瑞氏染液固定染色15min。随机于镜下取上、下、左、右、中心5个视野,计数穿膜细胞数,取5个视野的总数表示肿瘤细胞的侵袭能力。每种细胞分MSP2nm刺激与不刺激两组,平行实验2次。
1.1.6统计学方法采用spss10.0统计学软件的配 对 资料t检验。设P<0.05为差异有统计学意义。
2结果
2.1 Western Blotting结果见图3。
图3western blot检测RONΔ160的表达
由图1可见,Western Blotting检测到3T3-R0NΔ160细胞株中RONΔ160蛋白的表达,而在NIH-3T3和载体对照3T3-pDR2细胞中没有表达。
2.2免疫沉淀实验结果见图4。
由图2可见,R0NΔ160在无MSP刺激时有自身酪氨酸磷酸化,在MSP刺激下酪氨酸磷酸化略有增强,而空白对照和载体对照均未检测到R0NΔ160蛋白酪氨酸磷酸化表达。
2.3细胞形态变化见封二图5。
由图5可见,3T3细胞转染并表达R0NΔ160后,在形态上有明显变化:细胞较小,呈梭形或短棒状,细胞间紧密,成簇生长。
2.4 软琼脂培养基生长实验结果见图6A,封二图6B。
由图4可见,经过统计,3T3-RONΔ160细胞生长克隆数为146.00±3.61(数据均为MSP刺激组),而未转染组NIH-3T3细胞和载体对照组3T3-pDR2细胞分别为11.67±1.15和12.67±2.52,转染组与未转染组、载体对照组比较,差异均有统计学意义(t分别=51.60、151.19,P均<0.05)。
2.5体外跨室趋化运动实验结果见图7A、封二图7B。
由图5可见,转染组3T3-RONΔ160细胞穿膜细胞数为458.50±4.95(数据均为MSP刺激组),而未转染组NIH-3T3细胞和载体对照组3T3-pDR2细胞分别为20.00±5.66和21.50±4.95,转染组与未转染组、载体对照组比较,差异均有统计学意义(t分别=437.00、58.47,P均<0.05)。
3讨论
受体型酪氨酸激酶RON是属于MET原癌基因家族的成员,最早在一株转化的人类包皮角化细胞中被克隆 [1] 。RON的配体是MSP [2] ,也被称为肝细胞生长因子样蛋白(hepatocyte growth factor-like pro-tein)。RON被发现在一些乳癌、肺癌、膀胱癌和大肠癌等多种恶性肿瘤细胞株中有高表达并有报道发现高表达的RON在肿瘤转移中起重要作用 [4,5] 。酪氨酸激酶功能域的点突变可诱导RON的致肿瘤性 [6] 。mRNA剪切往往也可以导致其所编码的蛋白中大片断氨基酸序列的缺失 [7] 。在一些其他的肿瘤蛋白,如HER2、Smad2、MET中,也有因mRNA剪切而形成变异体的报道。RON有多个天然变异体,包括RONΔ165、RONΔ160、RONΔ155及sf-RON等,都被认为是RON的剪切变异体 [8,9] 。不同的RON变异体在不同的外显子缺失时表现出自身酪氨酸磷酸化,而使表达该变异体的细胞表现出转化细胞和肿瘤细胞的特性。RONΔ160是野生型RONmRNA的第5、6外显子剪切后表达的变异蛋白,最早在2000年从结肠 注: 软琼脂培养基生长实验中,3T3-RONΔ160细胞与3T3
及3T3-pDR2细胞比较,克隆生长数有显著增多,细癌细胞株HT-29中被克隆并初步揭示了其基本特性 [4] 。
NIH-3T3细胞是小鼠来源的成纤维细胞株。由于它显示出强烈的接触抑制作用,所以能明确地识别已发生转化的细胞,并作为一种重要的细胞材料使用于由肿瘤病毒和致癌剂等所引起的体外致癌研究。在本实验中,RONΔ160转染到NIH-3T3细胞中,建立稳定表达RONΔ160蛋白的3T3-RONΔ160细胞株。本次研究在免疫沉淀实验中,使用酪氨酸磷酸化抗体沉淀蛋白后,用RON特异性抗体行western blot检测,发现RONΔ160蛋白在没有MSP刺激时也有强酪氨酸磷酸化,这说明RONΔ160因胞外域中5、6外显子的剪切丢失,可能使蛋白结构发生变化而具有自身酪氨酸磷酸化能力;在MSP刺激下RONΔ160的酪氨酸磷酸化略有增强,说明RONΔ160保持了与MSP反应的能力,使配体MSP刺激途径也可以加强其酪氨酸磷酸化。
3T3-RONΔ160细胞与NIH-3T3细胞相比,细胞形态明显变化:细胞缩小,细胞角变少,呈短棒状,细胞间隙变小,成簇生长,接触抑制减弱。3T3-RONΔ160细胞在软琼脂培养基中的克隆生长数比NIH-3T3细胞和载体对照3T3-pDR2细胞高10倍以上。这些形态的变化和软琼脂基生长能力的大大增强有力地说明3T3-RONΔ160细胞具有明显的转化细胞特性。在排除载体影响后,RONΔ160的转染和表达成为唯一的原因。体外跨室趋化运动能力可以表示一种细胞的运动能力或者是肿瘤细胞的浸润能力。在本研究中,3T3-RONΔ160细胞表现出比阴性对照和载体对照高20倍以上的跨膜运动能力,充分表现出了RONΔ160蛋白的表达对NIH-3T3细胞的转化能力和肿瘤化能力。RONΔ160对细胞形态及细胞运动能力的作用与RON的酪氨酸激酶功能域的点突变相似 [6] 。
虽然本次研究对RONΔ160的基本特性有了一 定的了解,但是在具体的分子信号途径、体内的致瘤性以及RONΔ160是否能成为抗肿瘤的一个重要靶向基因等更多问题上还需做进一步研究。
【】
1 Ronsin C,Muscatelli F,Mattei MG,et al.A novel putativereceptor proteintyrosine kinase ofthemetfamily[J].Oncogene,1993,8(5):1195-1202.
2GaudinoG,Follenzi A,NaldiniL,etal.RONis a heterodimerictyrosinekinasereceptoractivatedbytheHGFhomologueMSP[J].EMBO J,1994,13(15):3524 3532.
3 Wang MH,Kurtz AL,Chen YQ.Identification of a novelsplicing product ofthe RON receptor tyrosine kinase in human colorectal carcinoma cells[J].Carcinogenesis,2000,21(8): 1507-1512.
4ZinserGM,LeonisMA,ToneyK,et al.Mammary-specificRonreceptoroverexpressioninduceshighlymetastaticmammarytumorsassociatedwithbeta-cateninactivation[J].CancerRes,2006,66(24):11967-11974.
5Hsu PY,Liu HS,ChengHL,et al.Collaboration of RON andepidermal growthfactorreceptor inhuman bladder carcinogenesis[J].JUrol,2006,176(5):2262-2267.
6Santoro M,PenengoL,Minetto M,etal.Pointmutationsinthetyrosinekinasedomainreleasetheoncogenic andmetastaticpoten- tialoftheRonreceptor[J].Oncogene,1998,17(6):741-749.
7 Misteli T,Spector DL.The cellular organization ofgene expres- sion[J].CurrOpinCellBiol,1998,10(3):323-331.
8Wang MH,Wang D,ChenYQ.Oncogenicandinvasivepotentials of human macrophage-stimulating protein receptor,the RON receptor tyrosine kinase[J].Carcinogenesis,2003,23(8):1291 -1300.
9ChiaraB,BarbaraC,PieraM,etal.TruncatedRONtyrosinekinasedrives tumor cell progression and abrogates cell-cell adhesion through E-cadherin transcriptional repression[J].Cancer Re- search,2004,64(15):5154-5161.