Crick 4*4*4 遗传密码表是完全错误的----C、H、O、N、P、S 所构物质分子彼此完全加成反应通用原则的发现
来源:岁月联盟
时间:2010-07-12
43遗传密码表;排列数计算;错误; Abstract : This paper mainly reports the series of error existing in the 43 table of genetic codes given by F. H. C. Crick as following show:1.The 64 permutations of Adenosine , Guanosine , Cytidine , and Uridine versus 20 different amino acids is a big mathematical error for there is no necessary element ~permutation relationship between nucleotides (or nucleosides ) and amino acids. 2.If selected three nucleotides from four as Adenosine , Guanosine , Cytidine , and Uridine , then the total number of permutations are not only 64 . At the same time , that 64 permutations linearly connects to each other do not express the real spacial DNA molecule and mRNA molecule , for real DNA chain and mRNA chain themselves have already been the spacial permutations on different carbon atoms , different hydrogen atoms , different oxygen atoms , different nitrogen atoms , and different phosphor atoms ; and that 61 amino acids and 3 terminal signal molecules linearly connects together do not express the real spacial protein molecule , for the real protein molecule itself has already been the spacial permutation on different carbon atoms , different hydrogen atoms , different oxygen atoms , different nitrogen atoms , and different sulfur atoms.3. The 43 table of genetic codes is in fact that 64 segments of spacial room in real DNA molecule versus a real molecule of amino acids in protein , which is an obvious logical error . 4.Each relevant experiment proved the table of genetic codes has its own permutable pattern from chemical reactants to products , instead of the descriptions in the table of genetic codes .5.The application of the genetic codes is another kind of permutable calculation . From DNA chain to mRNA chain , t RNA , ribosome , peptides , and to protein , each step has its own permutable calculation from chemical reactants to products . None step has an indispensable relationship 2004年4月,笔者在化学会24届学术年会上以墙报形式展出了《C、H、O、N、P、S所构物质分子彼此加成反应的通用原则》[1] 一文,在讨论该加成反应通式对生物大分子反应的意义时,本人提出了对当前国内外基因工程、遗传学、生物化学、植物生等多个领域正在通用的Crick (1969) 所排43 遗传密码表的怀疑,引起与会专家的兴趣,现就本人的这一工作进行详细汇报。 引 言 1954年2月,美国物理学家Gamow根据Watson 和Crick发表的DNA双股螺旋结构,提出了DNA的腺嘌呤N5C5H5 ,鸟嘌呤N5C5H5 O,胞嘧啶N3C4H5O和胸腺嘧啶N2C5H6O2等四种碱基可能就是密码子的最初设想。1955-1956年,Gamow 陆续发表文章,从排列组合计算,1种碱基对应1种氨基酸不够,2种碱基的16种组合对应20种氨基酸也不够,4种碱基的256种组合对应20种氨基酸太多,只有三种碱基组成64种组合对应20种氨基酸较合适。1959年,Crick本人提出“中心法则”支持Gamow的假说;1961年,Crick 和Brenner用实验证明了细菌和噬菌体遗传密码的三联性质。1961年夏天,Nirenberg领导的生化小组合成了碱基尿嘧啶,然后用3个尿嘧啶合成了苯丙氨酸分子 [2],从而确定了Crick所排遗传密码表的第一个密码子的意义:三个尿嘧啶是一个苯丙氨酸的密码子,并由此拉开了实验室里反应发生结果论证Gamow所提四种碱基分子排列对应蛋白质的二十种氨基酸分子的排列数计算的序幕。1964年,威斯康星大学的Khorana合成出了一个UG交替的共聚物…UGUGUGUGUG…,并用之作为合成蛋白质的信使,产生了半胱氨酸和缬氨酸交替的多肽链…半胱氨酸-缬氨酸-半胱氨酸-缬氨酸…,由此得出“UGU是半胱氨酸的密码子和GUG是缬氨酸的密码子”结论,并首创了实验室里“DNA链上碱基顺序不同致使反应发生的结果不同”分辨Gamow 和Crick数学排列表中“某一类元素相同但顺序不同致使排列不同”的方法。1965-66,剑桥MRC分子生物学实验室的Clark等做出起始密码子结论;同一实验室的Brenner等和美国耶鲁大学的A.Garen等各自做出终止密码子结论。到1966年,关于Gamow所提出的64个排列对应20种氨基酸分子的遗传密码意义全部被实验室里的反应所破译。Crick在该系列反应的基础上排出了mRNA上的遗传密码表(1969)。 43 遗传密码表的排出被认为是20世纪人类史上的大事 [2],被认为是生物学上的元素周期表 [2] 。Nirenberg、Khorana和Holley等因出色地做出了与该密码有关的系列实验,共获了1968年诺贝尔生理学和医学奖(Crick本人因其1953年发表的DNA双股螺旋结构,已分获了1962年的诺贝尔医学和生理学奖)。就当前来看,Crick的43 遗传密码表仍是基因工程、细胞生物学、分子生物学、生物化学、生物遗传学等多个学术领域的核心内容之一。 一、Crick 43 遗传密码表总的错误 以1、2、3、4构成多少个三位数和广州、上海、北京三地有多少种直达火车票的排列数计算为例,排列数计算在具体应用中是显性排列(1、2、3、4组成的三位数的排列43;广州、上海、北京三地间直达火车票的排列P32)截取或挑选隐性排列(1、2、3、4、5、6、7、8、9构成的三位数的排列93;广州、上海、北京、天津等n个地点间直达火车票的排列Pn2 ),不是一个排列数概念盲目对应一个非排列数概念,所以一个正确的排列数应用计算包含隐性排列(应用对象,如“三位数”“直达火车票”自身的排列)和显性排列(给定元素针对应用对象的排列,如1、2、3、4针对三位数;北京、上海、广州针对直达火车票)两个性质完全相同的排列,且隐性排列数总不小于显性排列数。在排列数应用中,当隐性排列数小于显性排列数时,不能进行排列数应用计算;当隐性排列(即应用对象)不存在排列数模型时,除非明确规定其排列数模型,否则排列数应用完全错误。 Gamow (1954)和Crick(1968)将DNA链上的腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2四种碱基分子排列对应蛋白质的甘氨酸NC2H5O2、丝氨酸NC3H7O3等二十种氨基酸分子,是个典型的没有明确排列数应用对象(64个字母三联体排列是针对“氨基酸分子”?还是针对“蛋白质分子”?)和没有规定应用对象的排列数模型(“氨基酸分子”的排列数模型是什么?或者,“蛋白蛋分子”的排列数模型是什么?)的完全错误的排列数应用案例。若明确“氨基酸分子”做隐性排列(排列数应用对象),则隐性排列可规定为具体氨基酸分子关于C、H、O、N原子的平面内单一方向上的排列,但化学上没有氨基酸分子关于C、H、O、N原子的排列模型,Gamow和Crick也都回避了氨基酸分子关于C、H、O、N原子的排列模型问题,所以,不能进行排列数应用;若明确“蛋白质分子”做隐性排列(排列数应用对象),则隐性排列可规定为具体蛋白质分子关于甘NC2H5O2、丝NC3H7O3等蛋白质单位的平面内单一方向上的排列,但有机化学上也没有蛋白质分子关于甘NC2H5O2、丝NC3H7O3等蛋白质单位的排列数模型,Gamow和Crick又都回避了蛋白质分子关于甘NC2H5O2、丝NC3H7O3等蛋白质单位的排列数问题,所以,也不能用排列数知识进行计算。从Crick所排43 遗传密码表64个排列对应64个氨基酸分子(虽然有3个是始终信号)的即成事实看,Gamow 和Crick根据氨基酸分子个数20(隐性排列的个数)做出对碱基个数4(显性排列的元素个数)选三排列(43≈20,且43>20),得出64个密码子的结论,就像在回答1、2、3、4四个数组成多少个三位数时,根据三位数100到999的总数900(隐性排列的个数)做出对1、2、3、4等4个数(显性排列的元素数)选五排列(45≈900,且45>900)的计算,从而得出1、2、3、4四个数组成的三位数有1024个的结论!不但结论完全错误,而且排列数计算步骤荒唐,所用方法令人费解。比如:设某射击场常规训练时有四名射击手和20个不同靶子,若教练要求三个射击手同时射击同一靶子进行常规训练,也许大家不会说什么;但若教练要求三个射击手按照6个不同的排列顺序分射6个不同靶子和四个射击手选三按照24个不同的排列顺序分射20个不同靶子(4个靶子重复使用)进行常规训练,那么大家会说这位教练“有病”! 二、43 遗传密码表的逻辑错误 例样:某机车学校由n层高的摩天大楼组成,每个楼层设置一个专业,共有制造、驾驶、维修、服务等四类专业;另有某列火车由m节车厢组成,每个车厢设置一个颜色,共有赤、橙等20种不同的颜色。现规定,该机车学校全权负责该列火车的制造和运营。 比较发现:Crick所排43 遗传密码表中的单个有序碱基三联体对应一个氨基酸分子,相当于例样中连续三层楼房制造和运营列车的一列车厢,不是连续三层楼房内的三个专业的师生制造和运营一节车厢(若用机车学校师生的话,对四种不同专业的师生进行组合就行了,没必要对不同专业进行排列)。密码表应用中DNA链上全部碱基翻译为蛋白质链上的全部氨基酸单位,相当于样例中机车学校的摩天大楼(n层高)制造了一列火车(m节),而不是摩天大楼内3×(n÷4)×( 4÷3)个专业的师生制造了这列列车,且机车学校摩天大楼所制造的这列火车与摩天大楼的楼高有关,即n : m=3 : 1(Fisers等1972报道过MS2整个外壳蛋白基因的碱基排列顺序,有一个起始密码GUG和两个终止密码UAA,UAG,中间是用来编码外壳蛋白的387个碱基,这个数目同外壳蛋白质实际含有的129个氨基酸数目(387/3=129)相吻合)。所以,Crick所排43 遗传密码表的全部逻辑错误在于:对单个碱基三联体的氨基酸意义解释是,用一个物体所在空间位置(某三核苷酸段在DNA长链上的相对位置)去解释另一个空间位置所包含的物体(蛋白质长链的一个相对位置含有什么样的氨基酸单位);对完整DNA链的蛋白质意义解释是,用一个系列物体所在系列空间位置(系列碱基在DNA链上的系列相对位置)去解释另一个系列位置所包含的系列物体(蛋白质链上的一个个相对位置包含一个个氨基酸分子)。(这相当于渔翁在青海湖边钓出杭州西湖的鱼。) 具体分析如下: 1.Crick的43 密码表中的任一个字母三排列,不代表生物学意义上的腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2真实分子的任何构造和构象,只代表A、G、C、T四个字母在平面内单一方向上的一个选三排列。对于真实DNA分子而言,沿5′→3′或3′→5′方向阅读时,任意相邻三碱基真实分子在r维空间的任意一个构造和构象均可视为它们在r维空间的一个排列。所以,43密码表中,任意一个字母三联体相对于DNA分子的意义是:它表达DNA单链上其字母所指三个核苷酸分子段的相对空间位置,而不表达DNA链上的三个相应碱基分子。正像机车学校的摩天大楼上的任意相连三层楼房所占空间不会制造火车的一个车厢一样,没有了核苷酸分子的DNA链上空间不能够生出完整而真实的氨基酸分子!所以,一个字母三联体对应一个真实的氨基酸分子是错误的。 2.全部64个字母三联体都不代表生物学意义的腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2真实分子的任何构象和构造,都只代表48个A、48个G、48个C、48个T等192个字母在平面内单一方向上的64类三三排列(实际排列数将远远大于64)。对于真实DNA分子而言,沿5′→3′或3′→5′方向阅读时,连续192个碱基在r维空间的任意一个构造和构象都可视为该192个碱基分子在r维空间的一个排列。所以,DNA分子本身的构造和构象已经是DNA分子关于腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2在r维空间的真实排列,Crick所排密码表中64个字母三联体首尾相连相对于DNA分子而言,它仅仅表达DNA链上连续192个碱基分子共享的那段儿空间位置,而且这段儿空间位置抽象化后与一个192层高的摩天大楼所占空间位置没有什么区别,两者完全一样!所以,64个字母三联体对应64个真实的氨基酸分子(含密码表应用过程中,做为特例的“终止信号”对应色氨酸或精氨酸)是逻辑错误的。 3.逻辑定义上的混乱 离开Crick 43 密码表的DNA分子基础和蛋白质分子基础,则Crick 43 密码表可以看作是纯粹的A、G、C、U四个字母的64个排列及对其的相应64个定义(人为的规定),我们发现,Crick对排列的定义也是逻辑混乱的: (1)对于多个给定排列而言,根据它们的元素相同,但元素顺序不同的情况,将不同排列定义为不同的名字是可以理解的。(正确的定义) 如(U,A,C)→UAC, CAU, ACU, UCA, CUA, AUC 酪 组 苏 丝 亮 异亮 (C,G,A)→ CGA, GAC, ACG, CAG, GCA, AGC 精 天冬 苏 谷酰 丙 甘 然而同时,又将元素不同的排列定义为同一个名字,则势必引起定义上的混乱。(错误的定义): 如(U,A,C)→(UAC, CAU, ACU, UCA, CUA, AUC) UAU 酪 组 苏 丝 亮 异亮 酪? (C,G,A)→ (CGA, GAC, ACG, CAG, GCA, AGC) CGC 精 天冬 苏 谷酰 丙 甘 精? (2)密码子氨基酸意义的简并性,实际上是把不同的排列定义为相同的名字。对于排列数来讲,这实际上就是一种错误定义。 如:GUU, GUC, GUA, GUG 缬 缬 缬 缬 又如:CCU, CCC, CCA, CCG 脯 脯 脯 脯 三、Crick 43 遗传密码表的数学错误 以圆周率π值发现意义的评章为案例,圆周率π值小数点后3000位数字由1、2、3、4、5、6、7、8、9等九种数字组成,对应的关于π值发现意义的英文评论文章由a、b、c等二十六种字母组成,用1、2、3、4、5、6、7、8、9选二所得81个排列简并对应26种英文字母,由此建立从π值发现到π值发现评论文章的信息传递密码表。由于⑴九种数字选二组成的81个二位数表达π值小数点后3000位数本身错误;⑵26种英文字母因九种数字的排列数81而使26种英文字母有限重复到81种英文字母,并表达π值发现意义的英文评论文章错误;⑶π值小数点后3000位数根据上述信息传递表所指示的专一字母,从小数点起向右阅读,翻译出相应的英文评论文章,因单词拼写错误、句子语法错误、全文前后逻辑混乱,因而是篇错误的文章!这篇文章不具有π值发现意义的评论性质。 比较发现,Crick的43遗传密码表有如下根本性数学错误: 1.用腺嘌呤N5C5H5(实为腺苷酸)、鸟嘌呤N5C5H5O(实为鸟苷酸)、胞嘧啶N3C4H5O(实为胞苷酸)、胸腺嘧啶N2C5H6O2(实为胸腺苷酸)四种碱基的64个排列中的任意多个首尾相连代替DNA单链关于n个该四种核苷酸分子交替相连或重复相连的真实结构,是把DNA单链关于四种核苷酸分子的4 n 排列数模型错误地用成43 排列数模型。如:小膜虫属的端粒DNA序列是串联重复50个以上的G G G G T T,这里单个的 G G G G T T联是A、G、C、T等4 6 排列数模型中的一个排列,而Crick的密码表把这个排列错误地描述成为A、G、C、T等43 排列数模型下 G G G 和 G G T 两个排列的首尾相连(正确的排列数描述应该是43·43);这里50个GGGGTT重复相连联是A、G、C、T等4300 排列数模型下的一个排列,而Crick密码表把这个排列错误地描述成为A、G、C、T等43 排列数模型下 G G G 、G T T 、G G T 、T G G 、 T T G 等6个排列重复出现100次并首尾相连(正确的排数描述应该是43· 43· 43…43,共100个43相乘)。当把腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2抽象为1、2、3、4四个数学点,把DNA分子的一条链抽象为平面内关于该1、2、3、4四个数学点组成的n位数,则DNA分子关于其四个碱基的排列数模型是4n ,或 41· 41· 41…41, 共n个41个相乘,n表示DNA链上碱基的个数,也可视为DNA的长度;不是43或41· 41· 41模型下64个三位数的任意多个首尾相连。 2.依据生命体DNA单链碱基顺序,按照Crick的43 遗传密码表所给三字母排列对应具体氨基酸分子的指示所合成出的唯一确定顺序的肽链或蛋白质分子(像样例中按照信息传递表把π值小数点后数字翻译成唯一顺序的文章一样),不是该生命体已成事实的肽链或蛋白质分子!相对于该生命体的事实蛋白质分子或肽链,这条由密码表指导合成的蛋白质分子或肽链是逻辑混乱、方法古怪、不具遗传特性的蛋白质分子或肽链(像样例中的密码所写文章一样!),是一个错误的蛋白质分子或错误的肽链!具体地说:⑴ 除了抗菌素是简单肽链外,较复杂的蛋白质如酶、膜蛋白、结构蛋白、收缩蛋白、病毒蛋白等至少具有三级结构以至四级结构 [3] ,而43 密码表指导合成的蛋白质分子或肽链是一级结构(平面内单一方向性)。⑵没有任何证据表明生命体中DNA分子的相对长度(碱基个数)是蛋白质分子的相对长度(氨基酸残基个数)的3倍。⑶ DNA分子依据4 3 密码表合成蛋白质分子的唯一性,违背生命体既成蛋白质的多种性和多态性。⑷密码表指导合成的蛋白质分子或肽链在平面内单一方向上的20 n/3 (n表示DNA链上碱基数)排列数模型不代表任何真实蛋白质分子在其r 维空间的排列数模型(蛋白质分子在r 维空间的真实排列是它的关于成份原子的空间构造)。⑸密码表指导合成的蛋白质分子或肽链上的起点和终点,在生命体既成蛋白质分子或肽链上不存在。⑹没有任可证据表明天然蛋白质完全水解后,其氨基酸分子的一级结构恰好同于且只同于DNA单链据43 密码表所翻译而成的肽链!⑺密码表中排列数对应自身的逻辑对等,允许使用天然蛋白质的氨基酸顺序反推相应DNA的碱基顺序。 3.腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2四个真实分子选三个真实分子进行排列计算上的错误。 核酸分子在细胞质中以可溶形式存在,所以具有布朗运动和丁达尔现象等分子的共同性质。真实的DNA碱基分子在n维空间的溶液中独立存在时,单个腺嘌呤N5C5H5相当于n维向量A: A=(A1,A2,A3…An);单个鸟嘌呤N5C5H5O相当于n维向量 G: G=( G1,G2,G3…Gn) ; 单个胞嘧啶N3C4H5O相当于n维向量C: C=( C1,C2,C3…Cn);单个胸腺嘧啶N2C5H6O2 相当于n维向量T: T=( T1,T2,T3…Tn)。依Gamow 和Crick选三排列的需要,我们令n维向量A、n维向量 G、n维向量C、n维向量T三三相乘,得其向量相乘的内积如下: [A,G,C]=A1G1C1+ A2G2C2+ A3G3C3+…+AnGnCn; [G,C,T]= G1C1T1+ G2C2T2+ G3C3T3+…+GnCnTn; [A,C,T]= A1C1T1+ A2C2T2+ A3C3T3+…+AnCnTn; [A,G,T]= A1G1T1+ A2G2T2+ A3G3T3+…+AnGnTn; 这里4n个内积的乘法项都可视为四个碱基分子在真实环境中的三排列,从具体的乘法项看,其存在与否只与四个向量所在的维数和在该维数上的分量有关,与其在该维上的顺序无关;从总的乘法项数目看,总排列数4n与Crick密码表中的个数64无关。 根据四种碱基分子在纸平面内的分子式(非结构式)N5C5H5、N5C5H5O、N3C4H5O、N2C5H6O2 可以分别出写出腺嘌呤、鸟嘌呤、胞嘧啶、胸腺嘧啶四种碱基分子的不完整m×n矩阵如下:
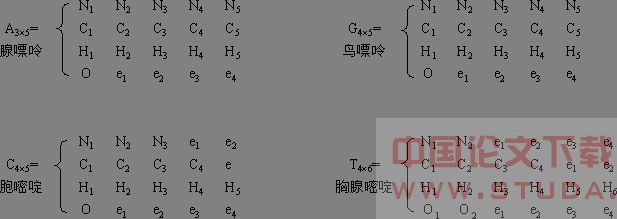 此时,由矩阵与向量组的关系 [4] 知,腺嘌呤 N5C5H5可以看作3个5维向量;鸟嘌呤N5C5H5O可以看作4个5维向量;胞嘧啶N3C4H5O可以看作4个5维向量;胸腺嘧啶N2C5H6O2可以看作 4个6维向量 ;这一情况下,无论用矩阵的三三相加,还是用矩阵的三三相乘,都无法得到 Crick 4 3 密码表中所用的关于四碱基的排列。 根据四种碱基分子在平面内的结构式,如下所示例样:
此时,由矩阵与向量组的关系 [4] 知,腺嘌呤 N5C5H5可以看作3个5维向量;鸟嘌呤N5C5H5O可以看作4个5维向量;胞嘧啶N3C4H5O可以看作4个5维向量;胸腺嘧啶N2C5H6O2可以看作 4个6维向量 ;这一情况下,无论用矩阵的三三相加,还是用矩阵的三三相乘,都无法得到 Crick 4 3 密码表中所用的关于四碱基的排列。 根据四种碱基分子在平面内的结构式,如下所示例样: 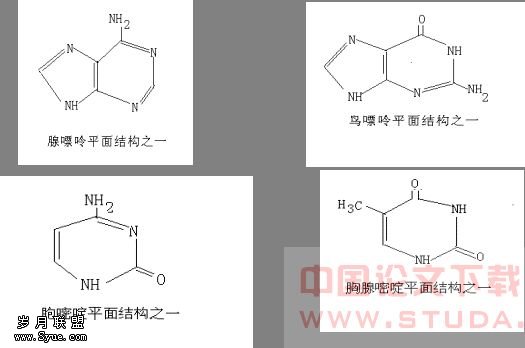
腺嘌呤N5C5H5, 鸟嘌呤N5C5H5O,胞嘧啶N3C4H5O,胸腺嘧啶N2C5H6O2四种真是分子的各种平面结构式和它们的互变异构体的各种平面结构式都可以看做其给定的平面几何图形,如下图所示:
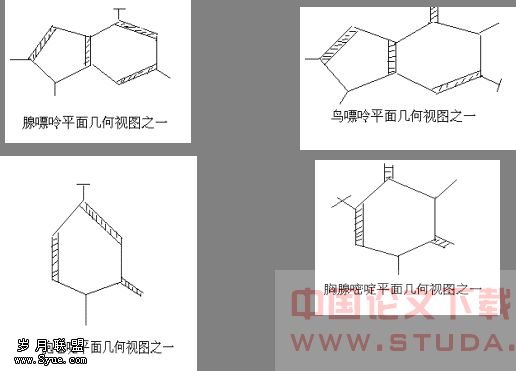 当对这些奇怪的平面几何图形进行严格的化学意义量化后,任选多个真实分子的平面结构式进行排列就可视作相关分子结构式的平面几何图形在平面内的拼图(比如 1 2 3 与 1 2 ε 不同; 7 6 6 与 L 6 6 不同)。 拼图数的多少可以近似表达这些分子结构式的平面排列数。根据Gamow和Crick要求,任选三个碱基分子的结构式并做它们的平面几何视图,无论是边对边拼图,还是角对角拼图,或角对边拼图,其拼图数都不是43 密码表中的排列数64。 设定化学反应中,所有参与反应的分子、原子、功能基团、离子和都不旋转、不振动、不发生物理位移,且在溶液中各自完全独立,则根据腺、鸟、胞、胸四碱基的分子式反映的原子成分,将各种分子视作各种集合如下:腺嘌呤 N5C5H5视作原子集合A: A={Na1,Na2,Na3,Na4,Na5, Ca1,Ca2,Ca3,Ca4,Ca5, Ha1,Ha2,Ha3,Ha4,Ha5;鸟嘌呤N5C5H5O视作原子集合G : G={Ng1,Ng2,Ng3,Ng4,Ng5,Cg1,Cg2,Cg3,Cg4,Cg5;Hg1,Hg2,Hg3,Hg4,Hg5,Og1;}; 胞嘧啶N3C4H5O视作原子集合C : C={Nc1 ,Nc2 ,Nc3 ,Cc1,Cc2,Cc3,Cc4 , Hc1,Hc2,Hc3,Hc4,Hc5,Oc};胸腺嘧啶N2C5H6O2视作原子集合T : T={Nt1,Nt2,Ct1,Ct2,Ct3,Ct4,Ct5,Ht1,Ht2,Ht3,Ht4,Ht5,Ht6,Ot1,Ot2}。这种情况下,通过集合间的相交和相并计算,可以得到Crick 43 密码表中各个氨基酸分子关于其密码子三碱基分子的原子的集合,但这类运算无法得到密码表中三碱基的有序集合联。 根据杨星阳关于C、H、O、N、P、S所构物质分子彼此完全加成反应的通用原则,发现核苷分子(不是碱基分子,也不是核苷酸分子)与氨基酸分子之间存在着如下关系: (1)1个腺苷分子 =5个甘氨酸分子 − 6H2O (2)1个尿苷分子 = 1个谷氨酸+1个谷氨酰胺 − 2H2O (3)1个胞苷分子 = 3个丝氨酸 分子 − 4H2O,等说明DNA的四种碱基分子与蛋白质的20种氨基酸分子之间存在着非常明确的数量和结构关系!这种关系在Crick 43 遗传密码表中根本没有体现出来。 综合这里的向量内积法、矩阵算法、分子构造式的平面图形拼图法、集合计算法等多种方法和杨星阳关于C、H、O、N、P、S所构物质分子彼此完全加成反应通用原则的应用,发现从真实腺、鸟、胞、胸四碱基分子中任选三个的排列数计算都不是43遗传密码表所引用的排列数64;所以,我们只能得出一种结论:Crick 43 遗传密码表所用的64个三三排列,有且只有对向量空间中真实分子腺嘌呤的代号字母A、真实分子鸟嘌呤的代号字母G、真实分子胞嘧啶的代号字母C、真实分子胸腺嘧啶的代号字母T 做平面内直线上的选三排列时才能成立!所以,这64个三三排列与真实腺嘌呤分子、真实鸟嘌呤分子、真实胞嘧啶分子、真实胸腺嘧啶分子无关!所以,Crick 43 密码表是远离化学反应的,远离化学家的实验室的!用化学家的反应实验来证明 43 密码表中各个排列的存在是错误的!四个独立碱基分子的64个一维线性排列何时存在,正如太阳、地球、月亮三者何时同时出现日食和月食需要天文学上的数学计算去证明那样,应该用分子运动论的数学方程去证明,不是用实验事件中反应能否发生和如何发生来证明。 另外,Crick 用mRNA的碱基代号A、G、C、U选三排列替换DNA的碱基代号A、G、C、T的选三排列时,没有对A、G、C、T、U五个字母作五选四的选择 C54,而直接地把“T”拿下,把“U”放上就完事了,这是不可思议的!这在排列数计算上是严重错误的。 四、实验体系不支持Crick 43 遗传密码表 为了论述上的方便,我们先设定Crick引用的各种实验体系支持其 43 密码表,然后看看排列数方法描述实验事件的后果是什么,从而得出密码表中所引用的各种已发生反应其生成物与反应物间关系均不符合排列数知识上的排列与元素间的关系。 (一)设定43密码表中各个字母是其所指的一个真实分子腺嘌呤N5C5H5、一个真实分子鸟嘌呤N5C5H5O、一个真实分子胞嘧啶N3C4H5O、一个真实分子尿嘧啶N2C4H4O2,且分别看时,每个字母联表示三个有序排列的独立碱基分子,而不是由三个核苷酸分子化合生成的一个分子。 (二)设定43 密码表中各个“字母三联体对应一个氨基酸分子”是指:正常生理条件下,字母三联体代表的真实分子(指三个碱基分子,不是指磷酸与该三个碱基分子组构而成的一个核苷酸分子),按平面内单一方向上的该顺序排列进行反应时,生成其所指示的1个氨基酸分子(不是别的氨基酸分子),且该反应机理可以表述为有限个基元反应,每个基元反应中生成物成份原子关于其反应物成份原子的平面内单一方向上的线性排列数模型存在。 (三)设定氨基酸分子合成肽链(蛋白质)技术的出现滞后于Gamow关于43 遗传密码表的提出,从而保证肽链关于氨基酸分子的排列不是关于溶液中“H+”、 “CH3-”等非氨基酸分子的排列,实现43密码表中64个“奇怪对应”(密码子的氨基酸意义)做为排列的各自独立。 那么,我们可以认为Crick 43 密码表相对于实验事件是正确的。这时,根据不同排列及其“破译实验”的事实,我们可以得出系列结论如下: (一)对于“UUU苯丙、GGG甘、CCC脯、AAA赖”四个密码子破译,可分别写出总反应式如下四个反应: U + U + U →苯丙 G + G + G →甘 C + C + C →脯 A + A + A →赖 由于在密码表中和在具体应用中,UUU不能写作 U,或 UU,UUUU,UUUUU等;GGG不能写作G ,或GG,或GGGG,GGGGG等,CCC不能写作C, 或CC, 或CCCC, CCCCC等,AAA不能写作A,或AA,或AAAA,AAAAA等,所以这四个反应都要求该类反应发生的机理是反应物与生成物在质量上的比率是3:1。而实际上这四个密码子的破译实验显然不是按照“3个同类分子完全化合得出其所指的1个氨基酸分子”进行反应的。 (二)(G,U,C)→(缬,丝,亮,精,丙,半胱),(A,U,G)→(异亮,丝,终止,终止,缬,天门冬),(A,C,U)→(苏,异亮,组,亮,酪,丝),(A,G,C)→(丝,苏,天门冬,丙,甘,精)等24个密码子的“破译实验”,可分别写出总反应式为如下的各类反应: G + U + C →缬 (反应1) A + U + G →异亮 (反应1) G + U + C →丝 (反应2) A + U + G →丝 (反应2) G + U + C →亮 (反应3) A + U + G →终止(色) 反应3) G + U + C →精 (反应4) A + U + G →终止(精) 反应4) G + U + C →丙 (反应5) A + U + G →缬 (反应5) G + U + C →半胱 (反应6) A + U + G →天门冬 (反应6) A + C + U →苏 (反应1) A + G + C →丝 (反应1) A + C + U →异亮 (反应2) A + G + C →苏 (反应2) A + C + U →组 (反应3) A + G + C →天门冬 (反应3) A + C + U →亮 (反应4) A + G + C →丙 (反应4) A + C + U →酪 (反应5) A + G + C →甘 (反应5) A + C + U →丝 (反应6) A + G + C →精 (反应6) 这里每一组反应中的6个反应实际上是当反应物为三种不同物质时,生成物种类数6恰好等于反应物a, b, c 的平面内单一方向上的全排列数P33,且反应物的一个线性排列恰好对应一个生成物,所以我们得出结论:从碱基分子到氨基酸分子的合成反应中,生成物种类数与反应物种类数间存在着平面内线性排列数对应关系,即生成物的种类数与反应物种类数的排列数相等,且生成物与反应物的平面内线性排列一一对应,由此得出两个推论。 推论一:由于生成物氨基酸总种类数是20,反应物DNA上碱基总种类数是4,所以从反应物到生成物之间存在着反应物种类数4与生成物种类数20的排列数对应关系:P44~(20 + 4个始终信号),由此我们推知另有不带简并性的四联体密码表的存在, 即A、G、C、U的全排列 =4×3×2×1 对应20种氨基酸分子和4个起始与终止信号,而这张P44 密码表的密码子四联性显然冲击Crick 密码表的三联唯一性,所以,该24个密码子的破译实验有漏洞。 推论二:由于
当对这些奇怪的平面几何图形进行严格的化学意义量化后,任选多个真实分子的平面结构式进行排列就可视作相关分子结构式的平面几何图形在平面内的拼图(比如 1 2 3 与 1 2 ε 不同; 7 6 6 与 L 6 6 不同)。 拼图数的多少可以近似表达这些分子结构式的平面排列数。根据Gamow和Crick要求,任选三个碱基分子的结构式并做它们的平面几何视图,无论是边对边拼图,还是角对角拼图,或角对边拼图,其拼图数都不是43 密码表中的排列数64。 设定化学反应中,所有参与反应的分子、原子、功能基团、离子和都不旋转、不振动、不发生物理位移,且在溶液中各自完全独立,则根据腺、鸟、胞、胸四碱基的分子式反映的原子成分,将各种分子视作各种集合如下:腺嘌呤 N5C5H5视作原子集合A: A={Na1,Na2,Na3,Na4,Na5, Ca1,Ca2,Ca3,Ca4,Ca5, Ha1,Ha2,Ha3,Ha4,Ha5;鸟嘌呤N5C5H5O视作原子集合G : G={Ng1,Ng2,Ng3,Ng4,Ng5,Cg1,Cg2,Cg3,Cg4,Cg5;Hg1,Hg2,Hg3,Hg4,Hg5,Og1;}; 胞嘧啶N3C4H5O视作原子集合C : C={Nc1 ,Nc2 ,Nc3 ,Cc1,Cc2,Cc3,Cc4 , Hc1,Hc2,Hc3,Hc4,Hc5,Oc};胸腺嘧啶N2C5H6O2视作原子集合T : T={Nt1,Nt2,Ct1,Ct2,Ct3,Ct4,Ct5,Ht1,Ht2,Ht3,Ht4,Ht5,Ht6,Ot1,Ot2}。这种情况下,通过集合间的相交和相并计算,可以得到Crick 43 密码表中各个氨基酸分子关于其密码子三碱基分子的原子的集合,但这类运算无法得到密码表中三碱基的有序集合联。 根据杨星阳关于C、H、O、N、P、S所构物质分子彼此完全加成反应的通用原则,发现核苷分子(不是碱基分子,也不是核苷酸分子)与氨基酸分子之间存在着如下关系: (1)1个腺苷分子 =5个甘氨酸分子 − 6H2O (2)1个尿苷分子 = 1个谷氨酸+1个谷氨酰胺 − 2H2O (3)1个胞苷分子 = 3个丝氨酸 分子 − 4H2O,等说明DNA的四种碱基分子与蛋白质的20种氨基酸分子之间存在着非常明确的数量和结构关系!这种关系在Crick 43 遗传密码表中根本没有体现出来。 综合这里的向量内积法、矩阵算法、分子构造式的平面图形拼图法、集合计算法等多种方法和杨星阳关于C、H、O、N、P、S所构物质分子彼此完全加成反应通用原则的应用,发现从真实腺、鸟、胞、胸四碱基分子中任选三个的排列数计算都不是43遗传密码表所引用的排列数64;所以,我们只能得出一种结论:Crick 43 遗传密码表所用的64个三三排列,有且只有对向量空间中真实分子腺嘌呤的代号字母A、真实分子鸟嘌呤的代号字母G、真实分子胞嘧啶的代号字母C、真实分子胸腺嘧啶的代号字母T 做平面内直线上的选三排列时才能成立!所以,这64个三三排列与真实腺嘌呤分子、真实鸟嘌呤分子、真实胞嘧啶分子、真实胸腺嘧啶分子无关!所以,Crick 43 密码表是远离化学反应的,远离化学家的实验室的!用化学家的反应实验来证明 43 密码表中各个排列的存在是错误的!四个独立碱基分子的64个一维线性排列何时存在,正如太阳、地球、月亮三者何时同时出现日食和月食需要天文学上的数学计算去证明那样,应该用分子运动论的数学方程去证明,不是用实验事件中反应能否发生和如何发生来证明。 另外,Crick 用mRNA的碱基代号A、G、C、U选三排列替换DNA的碱基代号A、G、C、T的选三排列时,没有对A、G、C、T、U五个字母作五选四的选择 C54,而直接地把“T”拿下,把“U”放上就完事了,这是不可思议的!这在排列数计算上是严重错误的。 四、实验体系不支持Crick 43 遗传密码表 为了论述上的方便,我们先设定Crick引用的各种实验体系支持其 43 密码表,然后看看排列数方法描述实验事件的后果是什么,从而得出密码表中所引用的各种已发生反应其生成物与反应物间关系均不符合排列数知识上的排列与元素间的关系。 (一)设定43密码表中各个字母是其所指的一个真实分子腺嘌呤N5C5H5、一个真实分子鸟嘌呤N5C5H5O、一个真实分子胞嘧啶N3C4H5O、一个真实分子尿嘧啶N2C4H4O2,且分别看时,每个字母联表示三个有序排列的独立碱基分子,而不是由三个核苷酸分子化合生成的一个分子。 (二)设定43 密码表中各个“字母三联体对应一个氨基酸分子”是指:正常生理条件下,字母三联体代表的真实分子(指三个碱基分子,不是指磷酸与该三个碱基分子组构而成的一个核苷酸分子),按平面内单一方向上的该顺序排列进行反应时,生成其所指示的1个氨基酸分子(不是别的氨基酸分子),且该反应机理可以表述为有限个基元反应,每个基元反应中生成物成份原子关于其反应物成份原子的平面内单一方向上的线性排列数模型存在。 (三)设定氨基酸分子合成肽链(蛋白质)技术的出现滞后于Gamow关于43 遗传密码表的提出,从而保证肽链关于氨基酸分子的排列不是关于溶液中“H+”、 “CH3-”等非氨基酸分子的排列,实现43密码表中64个“奇怪对应”(密码子的氨基酸意义)做为排列的各自独立。 那么,我们可以认为Crick 43 密码表相对于实验事件是正确的。这时,根据不同排列及其“破译实验”的事实,我们可以得出系列结论如下: (一)对于“UUU苯丙、GGG甘、CCC脯、AAA赖”四个密码子破译,可分别写出总反应式如下四个反应: U + U + U →苯丙 G + G + G →甘 C + C + C →脯 A + A + A →赖 由于在密码表中和在具体应用中,UUU不能写作 U,或 UU,UUUU,UUUUU等;GGG不能写作G ,或GG,或GGGG,GGGGG等,CCC不能写作C, 或CC, 或CCCC, CCCCC等,AAA不能写作A,或AA,或AAAA,AAAAA等,所以这四个反应都要求该类反应发生的机理是反应物与生成物在质量上的比率是3:1。而实际上这四个密码子的破译实验显然不是按照“3个同类分子完全化合得出其所指的1个氨基酸分子”进行反应的。 (二)(G,U,C)→(缬,丝,亮,精,丙,半胱),(A,U,G)→(异亮,丝,终止,终止,缬,天门冬),(A,C,U)→(苏,异亮,组,亮,酪,丝),(A,G,C)→(丝,苏,天门冬,丙,甘,精)等24个密码子的“破译实验”,可分别写出总反应式为如下的各类反应: G + U + C →缬 (反应1) A + U + G →异亮 (反应1) G + U + C →丝 (反应2) A + U + G →丝 (反应2) G + U + C →亮 (反应3) A + U + G →终止(色) 反应3) G + U + C →精 (反应4) A + U + G →终止(精) 反应4) G + U + C →丙 (反应5) A + U + G →缬 (反应5) G + U + C →半胱 (反应6) A + U + G →天门冬 (反应6) A + C + U →苏 (反应1) A + G + C →丝 (反应1) A + C + U →异亮 (反应2) A + G + C →苏 (反应2) A + C + U →组 (反应3) A + G + C →天门冬 (反应3) A + C + U →亮 (反应4) A + G + C →丙 (反应4) A + C + U →酪 (反应5) A + G + C →甘 (反应5) A + C + U →丝 (反应6) A + G + C →精 (反应6) 这里每一组反应中的6个反应实际上是当反应物为三种不同物质时,生成物种类数6恰好等于反应物a, b, c 的平面内单一方向上的全排列数P33,且反应物的一个线性排列恰好对应一个生成物,所以我们得出结论:从碱基分子到氨基酸分子的合成反应中,生成物种类数与反应物种类数间存在着平面内线性排列数对应关系,即生成物的种类数与反应物种类数的排列数相等,且生成物与反应物的平面内线性排列一一对应,由此得出两个推论。 推论一:由于生成物氨基酸总种类数是20,反应物DNA上碱基总种类数是4,所以从反应物到生成物之间存在着反应物种类数4与生成物种类数20的排列数对应关系:P44~(20 + 4个始终信号),由此我们推知另有不带简并性的四联体密码表的存在, 即A、G、C、U的全排列 =4×3×2×1 对应20种氨基酸分子和4个起始与终止信号,而这张P44 密码表的密码子四联性显然冲击Crick 密码表的三联唯一性,所以,该24个密码子的破译实验有漏洞。 推论二:由于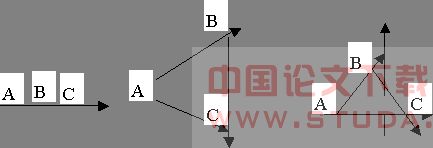 分属一维排列、三维排列、r维排列,空间维数不同,A,B,C的排列数不同;所以这里引述的每组反应数都是一维直线上的排列数;若把每组反应置于其真实r维空间,其三个反应物的排列数应大于其在r维空间的两两碰撞总次数。由于密码子破译实验的一切反应都是在r维空间进行,所以它们在r维空间的生成物种类数等于它们在r维空间的碰撞次数。— 而实际上,根据碰撞理论,并不是每次碰撞都能发生反应。碰撞频率中只有一部分能够发生反应。所以,密码表中针对排列数证明的实验事件是错误事件,或者说:对实验事件的排列数结论是错误结论! (三)UUG,AAC,UUC,UUA,GGC,GGA等36个有两重复元素的密码子的破译实验,连同UUU,AAA,GGG,CCC四个密码子的破译实验,可以分为(U,G)、(A,C)、(U,C)、(U,A)、(G,C)、(G,A)等6个系列,各个系列都可描述为如下的四类反应总式(表1-1)。如(U,C)→UUU 苯丙;CCC脯;UUC苯丙,CUU亮,UCU丝,CCU脯,表1-1: 对(X,Y)的选三重复排列(23=8)情况下生成物的种类数与反应物种类数的线性排列数相等。
分属一维排列、三维排列、r维排列,空间维数不同,A,B,C的排列数不同;所以这里引述的每组反应数都是一维直线上的排列数;若把每组反应置于其真实r维空间,其三个反应物的排列数应大于其在r维空间的两两碰撞总次数。由于密码子破译实验的一切反应都是在r维空间进行,所以它们在r维空间的生成物种类数等于它们在r维空间的碰撞次数。— 而实际上,根据碰撞理论,并不是每次碰撞都能发生反应。碰撞频率中只有一部分能够发生反应。所以,密码表中针对排列数证明的实验事件是错误事件,或者说:对实验事件的排列数结论是错误结论! (三)UUG,AAC,UUC,UUA,GGC,GGA等36个有两重复元素的密码子的破译实验,连同UUU,AAA,GGG,CCC四个密码子的破译实验,可以分为(U,G)、(A,C)、(U,C)、(U,A)、(G,C)、(G,A)等6个系列,各个系列都可描述为如下的四类反应总式(表1-1)。如(U,C)→UUU 苯丙;CCC脯;UUC苯丙,CUU亮,UCU丝,CCU脯,表1-1: 对(X,Y)的选三重复排列(23=8)情况下生成物的种类数与反应物种类数的线性排列数相等。 CUC亮,UCC丝;可以明显分为UUU苯丙对U元素三次重复,CCC脯对C 元素三次重复,UUC苯丙、CUU亮、UCU丝等对U二次重复,CCU脯、CUC亮、UCC丝等对C二次重复。各类反应中,生成物的种类数与反应物种类的平面内单一方向上的排列数相等。由此可得出结论:Crick 43 密码表中具有重复元素的密码子,在“氨基酸意义破译实验”中,同样具有生成物的种类数与反应物种类数的单向排列一一对应的关系。由此推论:对DNA的四种碱基中任二种碱基选n重复排列2n 情况下,生成物的种类数与反应物的种类数的单向排列数相等。将X,Y定义为两种碱基分子,规定n≤20,生成物1、2、3…n定义为具体的氨基酸分子(而不是肽链),则2n反应发生表同样是一个从DNA分子到蛋白质分子的遗传密码表。而这张2n遗传密码表的密码子n联性,直接与Crick 43 密码表的密码子三联性和三联唯一性相矛盾。— 所以,只能说 43 密码表中40个含重复碱基的密码子破译实验有问题,或者说,关于该类密码子的实验的描述方式错误。 (四)将43 密码表中全部192个碱基分子化学连接起来与64个(含起始与终止信号)氨基酸单位化学连接而成的肽链对应,可写出总反应式如下:U+U+U+A+U+G+…+G+G+G→Phe +Phe+ Ala +Leu+ …+Gly。从反应机制看,核苷酸链可视为由192个反应基组成的大分子,肽链可视为由64个反应基组成的大分子,而这个反应发生的原理是:反应物上的任意相邻三个反应基按照平面直线方向上的顺序决定生成一个生成物相应位置的基团,且非常专一。那么,我们得出结论:这样的反应,不是化学上讲的发生的反应,只能是人工制造的反应。我们知道,针对 43 遗传密码表中64个密码子的破译实验都是模拟生命条件下自然发生的实验,家们对生命遗传的化学反应推测也都是建立在自然发生反应的基础上进行推测,所以由密码表写出的总反应式永远不会在实验室里自然发生。关于密码表实验的总反应是错误反应。 综合这里的(一)、(二)、(三)、(四)推论,我们可以毫不犹豫地说,针对Crick 43遗传密码表的实验的排列数结论是错误的。那么,如此多的重大错误究竟出在实验的哪个环节呢?笔者认为,第一个环节出在Khorana将独立分子P、Q、R三者经 —“P”— “Q”—“R”— 分子链描述成为排列 P Q R ;第二个环节出在传统化学上反应发生实验的描述方式与数学上排列数知识两者的关系问题上。 具体地说,43 密码表中64个“密码子破译实验”,在反应式描述上有如下错误: (一)第一个密码子破译实验 U + m →苯丙氨酸,不能简单描述为 U + U + U + m →苯丙氨酸。Nirenberg(1969)在无细胞蛋白质合成体系中加入多聚尿苷酸合成出多聚苯丙氨酸的反应发生事件,没有给出尿嘧啶分子做为反应物有且只有三分子时,反应才发生的充分必要证明。况且,“UUU是苯丙氨酸的密码子”结论本身是在假设密码子是由三核苷酸联组成的设想下做出来的 [2] [5] [6]。 (二)将多聚腺苷酸和多聚胞苷酸分别注入加有14 C标记的不同氨基酸的培养液中,导致多聚赖氨酸和多聚脯氨酸的合成,在反应总式上描述为A+ m →赖 和C + m →脯 即可,不可描述为A + A + A→赖 和C + C + C →脯。前后两种描述方式在化学上的区别是:前者是泛指反应发生,后者是反应的量化要求,用杨星阳关于C、H、O、N、P、S所构物质分子加成反应通则(表达式)准确量化密码子破译实验中反应物与生成物间数量关系时,A + A +A ≠赖, C + C + C ≠脯。两种描述方式对 43 密码表的意义是:前者说明“遗传密码是单字母的”,后者说明“遗传密码是三字母的”。所以,AAA赖和 CCC 脯这两个密码子破译实验不成立。事实上,这两个密码子破译实验的排列数结论是效仿了Nirenberg“假设密码子是由三核苷酸链组成的”做法。 (三)“GGG甘”密码子的破译途径与“AAA赖、UUU苯丙、CCC脯”的不同 [6] 和肽链合成过程中“G + G + G→H+ 反应本质”共同表明,GGG甘的“破译实验”很可能是利用成熟的肽链合成技术,进行的实验上的投机行为。对于H2N---CH---COOH给定情况下,没有G、1个G、2个G、4个G 对于反应发生的真实环境来说,1个“H+ ”的获得都不是困难的事。所以,反应总式G + G + G →H+ 完全可以描述为G →H+ 或 G + G →H+,或G + G + G + G→H+。笔者认为,“GGG甘”密码子的破译实验是没有充分反证的“破译”。 (四)U 和 G的摩尔比为0.76和0.24的共聚体中UUU、UUG、UGU、GUU、UGG、GUG、GGU、GGG出现几率与以它们为模板时所测不同氨基酸的掺入率相比较“破译”相应8个半成品密码子实验 [6],如附表1-2和附表1-3所示,本质上是反应物不同量级上的光年级排列数(尿、鸟、胞、腺在不同分子个数上的选三排列)与生成物不同种类在质量多少上的排列顺序(不同氨基酸所占比率)一一对应,这一对应既不能说明密码子的三联唯一性,又是错误地把几率组合的空间多向性排列描述为平面内直线上的单向排列,所以不符合传统化学上对反应发生实验的描述方式,也严重缺乏排列数的周密性。另外,当用生成物(氨基酸分子)按比率配定一一对应反应物(碱基分子)时,Crick没有充分计算密码简并后同一种氨基酸分子在不同密码子上所占的比率,如苯丙∶亮∶异亮∶甘= 2∶6∶4∶4等。所以,这里比率配定的方法破译相应的半成品密码子排列的实验,相当于76只狼(或者白色金鱼)与24只羊(或者红色金鱼)在供有羊肉、麦草等8种食物的一个铁笼子里做共生实验,得到的结果是当2只狼与1只羊在一起时三者共食羊肉;当2只羊与1只狼在一起时,三者共食麦草。其他二核苷酸共聚体配定法所“破译”(U,A)8个密码子、(U,C)8个密码子、(A,C)8个密码子、(A,G)8个密码子和(C,G)8个密码子等实验的排列数结论缺陷与(U,G)相同。 (五)用Khorana所合成的有一定重复顺序的多核苷酸链“破译”密码子,犯的是另一类排列数错误,这涉及到化学反应上从反应物到生成物的排列数模型问题。(1)对于给定的P、Q、R三类不同核苷酸分子,当合成为PQRPQRPQR…PQR 共m个碱基构成的长链时,该链中的排列— P—Q—R—、— Q—R—P—、— R—P—Q—与Crick43遗传密码表中的排列PQR、QRP、RPQ大为不同。两者的本质区别在于前者是关于数学集合中元素(分子中的原子和功能基)的排列,后者是关于数学集合(整个分子)的排列。(2)当“…PQRPQRPQR…”为模板反应生出肽链“…aa1--aa1--aa1--aa1…”,“…QRPQRPQRP…”为模板反应生出肽链“…aa2--aa2--aa2--aa2…”,“…RPQRPQRPQ…”为模板反应生出肽链“…aa3--aa3--aa3--aa3…”时,为了迎合Gamow最初提出的43排列,把复杂的化学反应机理简单描述为PQR→氨基酸分子aa1 , QRP→氨基酸分子aa2, RPQ→氨基酸分子aa3,是将Crick 43遗传密码表中完整独立的氨基酸分子等同于其肽链合成中的反应基,如 — CH3 、—C2H5O、H+ 等,两者在数学排列模型上是完全不同的。(3)从单纯的氨基酸分子合成为肽链反应过程来看,相对于此前早已成熟的肽链合成技术,Khorana在肽链合成过程中掺入核苷酸分子链犹如“脱裤子放庇”。(4)从总反应式x (核苷酸链)→ y ( 肽链)看, Khorana的有序碱基链破译43 密码表中不同三联体字母顺序的方法,是用反应物的有限个反应基在平面直线上排列去规定生成物的一个反应基在平面直线上的具体位置。这一反应机制在自然发生的反应中是不存在的。(5)对于给定的P、Q、R三类不同碱基,当通过不同摩尔数配定比率的方法与Khorana的有重复顺序的多核苷酸链方法一起得出密码子的氨基酸意义破译结论时,只是简单地对反应结果做了归纳,而没有从反应机理上找到两者间在排列数模型上的对应通道,所以,两种方法混用的排列数结果并不可靠。(6)对“PQR PQR PQR PQR PQR”长链,Khorana不限定该链的长度,或从P读起,或从Q读起,或从R读起,实际上等于是把排列 123 与排列1231、排列12312视为相同;而将PQR PQR PQR PQR 长链作用生出aa1—aa1—aa1—aa1—aa1读作5个 PQR→aa1,无疑于是把花费123 123 123 123 123元钱买下的美国五角大楼,解读为5个“花费123元买下美国五角大楼的一角”。 (六)1964年,Nirenberg发现的“在无细胞蛋白质合成的情况下,三核苷酸能促进特异的t RNA分子与核糖体结合”,使得密码子破译实验从反应物到生成物的真实排列数模型一下子从原来的光年数量级演变成了光年数量级的光年数量倍。43 密码表在不考虑t RNA的发现引起密码表模型变化情况下,用该方法破译出大约50种密码子显然是回避化学分子关于其编号原子排列数模型的必然错误。 (七)各种实验体系中,针对DNA链的顺序问题和蛋白质链的顺序问题,都直接地把真实核苷酸分子长链关于不同核苷酸分子的空间构造简化为腺、鸟、胞、尿四个功能基团在平面内直线上的排列,而把真实肽链关于不同氨基酸单位的空间构造简化为“H-”、“CH3-”、“C3H7-”等20个侧基在平面内直线上的排列,等于是把 110 112 114 116 118 这类由10个“1”、1个“0”、1个“2”、1个“4”,1个“6”,1个“8”组成的615 个15位数(排列)一下子变成了 02468 这个由1个“0”、1个“2”、1个“4”、1个“6”、1个“8”共5个数组成的55 个5位数字(排列),排列数变少了,排列数的意义也变了。 综合本节各个论述点,可以看到针对Gamow 43遗传密码表的“密码子破译”实验事件,实际上是把实验室里腺嘌呤、鸟嘌呤、胞嘧啶、尿嘧啶四个真实的碱基分子和腺核苷酸、鸟核苷酸、胞核苷酸、尿核苷酸四个真实分子等同于Gamow所说的平面内A、G、C、U四个代号字母,利用实验上娴熟的合成肽链的技术,制造了多种体系的由DNA分子合成蛋白质(肽)链的反应发生事件。由于化学反应上,从反应物到生成物的排列模型没有建立,从编号原子到真实分子的排列数模型也没有建立,所以,所有反应发生事件在论证Gamow 的遗传密码表所排64个密码子过程中,都没有切实地用排列数方法对反应发生过程进行详细而正确的计算,而只是用生成物的氨基酸结果证明了这样的反应可以发生。所以,这样的实验是不证明43 排列的。 五、应用体系上的错误 43 遗传密码表应用体系上的全部错误在于:用一步(个)排列数计算替代无穷多步(个)排列数计算,通过步步(个个)错误的排列数计算(无穷多次显性排列错误的挑选相应的隐性排列),造成DNA链的固有信息步步失真描述,最后把DNA链的固有信息演变成了蛋白质链上甘、丝等具体氨基酸分子的信息。以排列数方法计算香港赵、钱、孙、李四大歌星的现场演唱会经发射塔、无线电信道、接收天线等多个步骤传递给北京20户人家的电视屏幕为例,考察发现Crick 所排43 遗传密码表在解决DNA分子和蛋白质分子间关系上的应用,首先是将DNA链的r维空间几何体(螺旋结构)未经排列数计算,失真表达为平面内直线上的排列结构;接下来又未经排列数计算,将DNA链平面内4m 模型的排列失真表达为43 模型下64个排列的首尾相连;转录时仍未经排列数计算,直接用mRNA的U替换DNA 的T,将平面内DNA的A、G、C、T的43 模型失真表达为mRNA的A、G、C、U的43 模型;翻译时,还未经排列数计算,直接用t RNA的氨基酸分子对应mRNA上的三核苷酸链,将mRNA的信息失真表达为t RNA上的氨基酸分子;肽链合成时,也未经排列数计算,将t RNA上的氨基酸分子信息失真表达为核糖体上某个具体的空间位置。就这样,通过步步失真表达(错误的排列数计算),将真实DNA链上的三核苷酸分子变成了蛋白质(肽)链上的一个空间位置;或者说,把DNA链上某三相邻核苷酸所在的位置演变成了蛋白质(肽)链上的某个空间位置上的具体氨基酸分子。这相当于样例中,香港四大歌星的现场演唱会用密码表的排列数方法传递到北京20户人家的电视屏幕时,经4大歌星的排列数与北京20台电视机一一对应的密码表指导造成步步失真传递后,变成了北京20户人家的电视机屏幕! 43 密码表在应用中的主要错误具体表现在以下几个方面:
CUC亮,UCC丝;可以明显分为UUU苯丙对U元素三次重复,CCC脯对C 元素三次重复,UUC苯丙、CUU亮、UCU丝等对U二次重复,CCU脯、CUC亮、UCC丝等对C二次重复。各类反应中,生成物的种类数与反应物种类的平面内单一方向上的排列数相等。由此可得出结论:Crick 43 密码表中具有重复元素的密码子,在“氨基酸意义破译实验”中,同样具有生成物的种类数与反应物种类数的单向排列一一对应的关系。由此推论:对DNA的四种碱基中任二种碱基选n重复排列2n 情况下,生成物的种类数与反应物的种类数的单向排列数相等。将X,Y定义为两种碱基分子,规定n≤20,生成物1、2、3…n定义为具体的氨基酸分子(而不是肽链),则2n反应发生表同样是一个从DNA分子到蛋白质分子的遗传密码表。而这张2n遗传密码表的密码子n联性,直接与Crick 43 密码表的密码子三联性和三联唯一性相矛盾。— 所以,只能说 43 密码表中40个含重复碱基的密码子破译实验有问题,或者说,关于该类密码子的实验的描述方式错误。 (四)将43 密码表中全部192个碱基分子化学连接起来与64个(含起始与终止信号)氨基酸单位化学连接而成的肽链对应,可写出总反应式如下:U+U+U+A+U+G+…+G+G+G→Phe +Phe+ Ala +Leu+ …+Gly。从反应机制看,核苷酸链可视为由192个反应基组成的大分子,肽链可视为由64个反应基组成的大分子,而这个反应发生的原理是:反应物上的任意相邻三个反应基按照平面直线方向上的顺序决定生成一个生成物相应位置的基团,且非常专一。那么,我们得出结论:这样的反应,不是化学上讲的发生的反应,只能是人工制造的反应。我们知道,针对 43 遗传密码表中64个密码子的破译实验都是模拟生命条件下自然发生的实验,家们对生命遗传的化学反应推测也都是建立在自然发生反应的基础上进行推测,所以由密码表写出的总反应式永远不会在实验室里自然发生。关于密码表实验的总反应是错误反应。 综合这里的(一)、(二)、(三)、(四)推论,我们可以毫不犹豫地说,针对Crick 43遗传密码表的实验的排列数结论是错误的。那么,如此多的重大错误究竟出在实验的哪个环节呢?笔者认为,第一个环节出在Khorana将独立分子P、Q、R三者经 —“P”— “Q”—“R”— 分子链描述成为排列 P Q R ;第二个环节出在传统化学上反应发生实验的描述方式与数学上排列数知识两者的关系问题上。 具体地说,43 密码表中64个“密码子破译实验”,在反应式描述上有如下错误: (一)第一个密码子破译实验 U + m →苯丙氨酸,不能简单描述为 U + U + U + m →苯丙氨酸。Nirenberg(1969)在无细胞蛋白质合成体系中加入多聚尿苷酸合成出多聚苯丙氨酸的反应发生事件,没有给出尿嘧啶分子做为反应物有且只有三分子时,反应才发生的充分必要证明。况且,“UUU是苯丙氨酸的密码子”结论本身是在假设密码子是由三核苷酸联组成的设想下做出来的 [2] [5] [6]。 (二)将多聚腺苷酸和多聚胞苷酸分别注入加有14 C标记的不同氨基酸的培养液中,导致多聚赖氨酸和多聚脯氨酸的合成,在反应总式上描述为A+ m →赖 和C + m →脯 即可,不可描述为A + A + A→赖 和C + C + C →脯。前后两种描述方式在化学上的区别是:前者是泛指反应发生,后者是反应的量化要求,用杨星阳关于C、H、O、N、P、S所构物质分子加成反应通则(表达式)准确量化密码子破译实验中反应物与生成物间数量关系时,A + A +A ≠赖, C + C + C ≠脯。两种描述方式对 43 密码表的意义是:前者说明“遗传密码是单字母的”,后者说明“遗传密码是三字母的”。所以,AAA赖和 CCC 脯这两个密码子破译实验不成立。事实上,这两个密码子破译实验的排列数结论是效仿了Nirenberg“假设密码子是由三核苷酸链组成的”做法。 (三)“GGG甘”密码子的破译途径与“AAA赖、UUU苯丙、CCC脯”的不同 [6] 和肽链合成过程中“G + G + G→H+ 反应本质”共同表明,GGG甘的“破译实验”很可能是利用成熟的肽链合成技术,进行的实验上的投机行为。对于H2N---CH---COOH给定情况下,没有G、1个G、2个G、4个G 对于反应发生的真实环境来说,1个“H+ ”的获得都不是困难的事。所以,反应总式G + G + G →H+ 完全可以描述为G →H+ 或 G + G →H+,或G + G + G + G→H+。笔者认为,“GGG甘”密码子的破译实验是没有充分反证的“破译”。 (四)U 和 G的摩尔比为0.76和0.24的共聚体中UUU、UUG、UGU、GUU、UGG、GUG、GGU、GGG出现几率与以它们为模板时所测不同氨基酸的掺入率相比较“破译”相应8个半成品密码子实验 [6],如附表1-2和附表1-3所示,本质上是反应物不同量级上的光年级排列数(尿、鸟、胞、腺在不同分子个数上的选三排列)与生成物不同种类在质量多少上的排列顺序(不同氨基酸所占比率)一一对应,这一对应既不能说明密码子的三联唯一性,又是错误地把几率组合的空间多向性排列描述为平面内直线上的单向排列,所以不符合传统化学上对反应发生实验的描述方式,也严重缺乏排列数的周密性。另外,当用生成物(氨基酸分子)按比率配定一一对应反应物(碱基分子)时,Crick没有充分计算密码简并后同一种氨基酸分子在不同密码子上所占的比率,如苯丙∶亮∶异亮∶甘= 2∶6∶4∶4等。所以,这里比率配定的方法破译相应的半成品密码子排列的实验,相当于76只狼(或者白色金鱼)与24只羊(或者红色金鱼)在供有羊肉、麦草等8种食物的一个铁笼子里做共生实验,得到的结果是当2只狼与1只羊在一起时三者共食羊肉;当2只羊与1只狼在一起时,三者共食麦草。其他二核苷酸共聚体配定法所“破译”(U,A)8个密码子、(U,C)8个密码子、(A,C)8个密码子、(A,G)8个密码子和(C,G)8个密码子等实验的排列数结论缺陷与(U,G)相同。 (五)用Khorana所合成的有一定重复顺序的多核苷酸链“破译”密码子,犯的是另一类排列数错误,这涉及到化学反应上从反应物到生成物的排列数模型问题。(1)对于给定的P、Q、R三类不同核苷酸分子,当合成为PQRPQRPQR…PQR 共m个碱基构成的长链时,该链中的排列— P—Q—R—、— Q—R—P—、— R—P—Q—与Crick43遗传密码表中的排列PQR、QRP、RPQ大为不同。两者的本质区别在于前者是关于数学集合中元素(分子中的原子和功能基)的排列,后者是关于数学集合(整个分子)的排列。(2)当“…PQRPQRPQR…”为模板反应生出肽链“…aa1--aa1--aa1--aa1…”,“…QRPQRPQRP…”为模板反应生出肽链“…aa2--aa2--aa2--aa2…”,“…RPQRPQRPQ…”为模板反应生出肽链“…aa3--aa3--aa3--aa3…”时,为了迎合Gamow最初提出的43排列,把复杂的化学反应机理简单描述为PQR→氨基酸分子aa1 , QRP→氨基酸分子aa2, RPQ→氨基酸分子aa3,是将Crick 43遗传密码表中完整独立的氨基酸分子等同于其肽链合成中的反应基,如 — CH3 、—C2H5O、H+ 等,两者在数学排列模型上是完全不同的。(3)从单纯的氨基酸分子合成为肽链反应过程来看,相对于此前早已成熟的肽链合成技术,Khorana在肽链合成过程中掺入核苷酸分子链犹如“脱裤子放庇”。(4)从总反应式x (核苷酸链)→ y ( 肽链)看, Khorana的有序碱基链破译43 密码表中不同三联体字母顺序的方法,是用反应物的有限个反应基在平面直线上排列去规定生成物的一个反应基在平面直线上的具体位置。这一反应机制在自然发生的反应中是不存在的。(5)对于给定的P、Q、R三类不同碱基,当通过不同摩尔数配定比率的方法与Khorana的有重复顺序的多核苷酸链方法一起得出密码子的氨基酸意义破译结论时,只是简单地对反应结果做了归纳,而没有从反应机理上找到两者间在排列数模型上的对应通道,所以,两种方法混用的排列数结果并不可靠。(6)对“PQR PQR PQR PQR PQR”长链,Khorana不限定该链的长度,或从P读起,或从Q读起,或从R读起,实际上等于是把排列 123 与排列1231、排列12312视为相同;而将PQR PQR PQR PQR 长链作用生出aa1—aa1—aa1—aa1—aa1读作5个 PQR→aa1,无疑于是把花费123 123 123 123 123元钱买下的美国五角大楼,解读为5个“花费123元买下美国五角大楼的一角”。 (六)1964年,Nirenberg发现的“在无细胞蛋白质合成的情况下,三核苷酸能促进特异的t RNA分子与核糖体结合”,使得密码子破译实验从反应物到生成物的真实排列数模型一下子从原来的光年数量级演变成了光年数量级的光年数量倍。43 密码表在不考虑t RNA的发现引起密码表模型变化情况下,用该方法破译出大约50种密码子显然是回避化学分子关于其编号原子排列数模型的必然错误。 (七)各种实验体系中,针对DNA链的顺序问题和蛋白质链的顺序问题,都直接地把真实核苷酸分子长链关于不同核苷酸分子的空间构造简化为腺、鸟、胞、尿四个功能基团在平面内直线上的排列,而把真实肽链关于不同氨基酸单位的空间构造简化为“H-”、“CH3-”、“C3H7-”等20个侧基在平面内直线上的排列,等于是把 110 112 114 116 118 这类由10个“1”、1个“0”、1个“2”、1个“4”,1个“6”,1个“8”组成的615 个15位数(排列)一下子变成了 02468 这个由1个“0”、1个“2”、1个“4”、1个“6”、1个“8”共5个数组成的55 个5位数字(排列),排列数变少了,排列数的意义也变了。 综合本节各个论述点,可以看到针对Gamow 43遗传密码表的“密码子破译”实验事件,实际上是把实验室里腺嘌呤、鸟嘌呤、胞嘧啶、尿嘧啶四个真实的碱基分子和腺核苷酸、鸟核苷酸、胞核苷酸、尿核苷酸四个真实分子等同于Gamow所说的平面内A、G、C、U四个代号字母,利用实验上娴熟的合成肽链的技术,制造了多种体系的由DNA分子合成蛋白质(肽)链的反应发生事件。由于化学反应上,从反应物到生成物的排列模型没有建立,从编号原子到真实分子的排列数模型也没有建立,所以,所有反应发生事件在论证Gamow 的遗传密码表所排64个密码子过程中,都没有切实地用排列数方法对反应发生过程进行详细而正确的计算,而只是用生成物的氨基酸结果证明了这样的反应可以发生。所以,这样的实验是不证明43 排列的。 五、应用体系上的错误 43 遗传密码表应用体系上的全部错误在于:用一步(个)排列数计算替代无穷多步(个)排列数计算,通过步步(个个)错误的排列数计算(无穷多次显性排列错误的挑选相应的隐性排列),造成DNA链的固有信息步步失真描述,最后把DNA链的固有信息演变成了蛋白质链上甘、丝等具体氨基酸分子的信息。以排列数方法计算香港赵、钱、孙、李四大歌星的现场演唱会经发射塔、无线电信道、接收天线等多个步骤传递给北京20户人家的电视屏幕为例,考察发现Crick 所排43 遗传密码表在解决DNA分子和蛋白质分子间关系上的应用,首先是将DNA链的r维空间几何体(螺旋结构)未经排列数计算,失真表达为平面内直线上的排列结构;接下来又未经排列数计算,将DNA链平面内4m 模型的排列失真表达为43 模型下64个排列的首尾相连;转录时仍未经排列数计算,直接用mRNA的U替换DNA 的T,将平面内DNA的A、G、C、T的43 模型失真表达为mRNA的A、G、C、U的43 模型;翻译时,还未经排列数计算,直接用t RNA的氨基酸分子对应mRNA上的三核苷酸链,将mRNA的信息失真表达为t RNA上的氨基酸分子;肽链合成时,也未经排列数计算,将t RNA上的氨基酸分子信息失真表达为核糖体上某个具体的空间位置。就这样,通过步步失真表达(错误的排列数计算),将真实DNA链上的三核苷酸分子变成了蛋白质(肽)链上的一个空间位置;或者说,把DNA链上某三相邻核苷酸所在的位置演变成了蛋白质(肽)链上的某个空间位置上的具体氨基酸分子。这相当于样例中,香港四大歌星的现场演唱会用密码表的排列数方法传递到北京20户人家的电视屏幕时,经4大歌星的排列数与北京20台电视机一一对应的密码表指导造成步步失真传递后,变成了北京20户人家的电视机屏幕! 43 密码表在应用中的主要错误具体表现在以下几个方面:(一)做为43 密码表应用的理论支撑的“中心法则”(Crick ,1971),如下图所示,有着自身独立的复杂的化学路径的排列数模型。
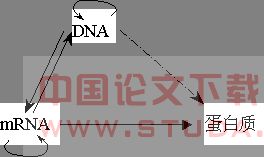 其模型的排列数模型完全背离43 密码表存在本身的“DNA→蛋白质”四选三排列模型。 (二)43 密码表中“字母三联体~氨基酸分子 ”是什么意思?“字母三联体~氨基酸分子”这种奇怪对应本身生物学意义模糊,对它的理解可谓是仁者见仁,智者见智。从该表产生的动机和该表的现状看,“奇怪对应”是指不考虑腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2四种碱基的原子成份、空间构造、物理性质情况下,把任意三个做为数学上的点进行排列后,对应同样不考虑原子成份、空间构造和物理性质的单个氨基酸分子。正是这样,保证了密码表的模型是43,密码子的总数是64;从针对该表的反应发生实验事件看,“奇怪对应”意指三字母所代表的三个真实核苷酸分子在充分考虑其原子成份、空间构造、物理性质和化学性质情况下,以直接参与形式或酶促合成形式导致针对具体氨基酸分子的反应发生。正是这样,各个实验事件的排列数模型都极其复杂,每存在一个反应发生实验,就存在一个从反应物原子排列到生成物原子排列的反应密码表。这些反应发生的密码表没有一个是43 的排列数模型;从该表的应用体系看,“奇怪对应”是指不考虑腺苷酸、鸟苷酸、胞苷酸、胸腺苷酸真实分子的原子成份、构造和理化性质,也不考虑甘氨酸、丙氨酸等全部氨基酸真实分子的原子成份、构造和理化性质,根据43 密码表的指示,直接地把DNA链上的三个相邻碱基变换成为三联体所指的氨基酸分子在蛋白质(肽)链上的一个空间位置。这三个方面各自对“字母三联体~氨基酸分子”的解释完全不一样!三者之间的排列数语言是不相通的。 (三)密码表中全部64个字母三联体都是平面内直线上的单向阅读,如AGU,不能读作UGA,前者是丝氨酸的密码子,后者是终止信号。而空间DNA双股螺旋单链上的字母三联体从5′→3′和从 3′→5′都是r维空间的多向阅读,如:
其模型的排列数模型完全背离43 密码表存在本身的“DNA→蛋白质”四选三排列模型。 (二)43 密码表中“字母三联体~氨基酸分子 ”是什么意思?“字母三联体~氨基酸分子”这种奇怪对应本身生物学意义模糊,对它的理解可谓是仁者见仁,智者见智。从该表产生的动机和该表的现状看,“奇怪对应”是指不考虑腺嘌呤N5C5H5、鸟嘌呤N5C5H5O、胞嘧啶N3C4H5O、胸腺嘧啶N2C5H6O2四种碱基的原子成份、空间构造、物理性质情况下,把任意三个做为数学上的点进行排列后,对应同样不考虑原子成份、空间构造和物理性质的单个氨基酸分子。正是这样,保证了密码表的模型是43,密码子的总数是64;从针对该表的反应发生实验事件看,“奇怪对应”意指三字母所代表的三个真实核苷酸分子在充分考虑其原子成份、空间构造、物理性质和化学性质情况下,以直接参与形式或酶促合成形式导致针对具体氨基酸分子的反应发生。正是这样,各个实验事件的排列数模型都极其复杂,每存在一个反应发生实验,就存在一个从反应物原子排列到生成物原子排列的反应密码表。这些反应发生的密码表没有一个是43 的排列数模型;从该表的应用体系看,“奇怪对应”是指不考虑腺苷酸、鸟苷酸、胞苷酸、胸腺苷酸真实分子的原子成份、构造和理化性质,也不考虑甘氨酸、丙氨酸等全部氨基酸真实分子的原子成份、构造和理化性质,根据43 密码表的指示,直接地把DNA链上的三个相邻碱基变换成为三联体所指的氨基酸分子在蛋白质(肽)链上的一个空间位置。这三个方面各自对“字母三联体~氨基酸分子”的解释完全不一样!三者之间的排列数语言是不相通的。 (三)密码表中全部64个字母三联体都是平面内直线上的单向阅读,如AGU,不能读作UGA,前者是丝氨酸的密码子,后者是终止信号。而空间DNA双股螺旋单链上的字母三联体从5′→3′和从 3′→5′都是r维空间的多向阅读,如: 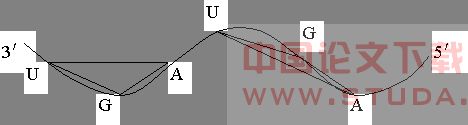
中的AGU、AGU各自都有三个不同的方向A→G、G→U、U→A。43 密码表的平面内直线上单向阅读的方法,强制阅读r维空间多向存在的DNA链上三邻碱基时,会发生同一个三邻碱基有两种读法(2个线性排列)和两个不同三邻碱基有一种相同读法(1个线性排列)的现象,这一现象对43 密码表应用的意义是:DNA上的一个三邻碱基理论上可以转录成两种不同的氨基酸分子;两个不同的三邻碱基理论上可以转录成同一种氨基酸分子。 (四)64个密码子各自独立、整体共存的排列数特性,要求按3′→5′或5′→3′方向阅读DNA链连续192个碱基时,对重复出现2次以上的三邻碱基进行跳读,从而保证密码应用中43 排列的切实存在。而实际应用中,很少是用跳读的方法进行转录的。如
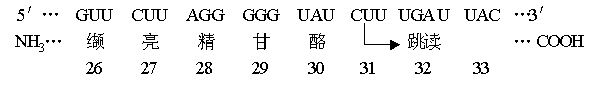 对第二个CUU应该跳读,实际应用中继续把第31个CUU读为亮氨酸。 (五)密码表应用性结论“高度重复序列和中度重复序列不编码蛋白质”与密码表引以确立的经典实验“PQRPQRPQR…PQR高度重复序列确立PQR→aa1,QRP→aa2 , RQP→aa3 , 三类密码子破译”完全相违背。这说明,Khorana合成的有确定顺序的核苷酸链用于破译密码子氨基酸意义的方法在具体应用中行不通。因而Khorana 的PQRPQRPQR…PQR法所确立的大量密码子的氨基酸意义是不准确的,是不可靠的。 (六) 若DNA链上某个三核苷酸分子段以分子模型形式独立存在,且在空间某域的相位不变,那么,观察者所站角度不同,该三核苷酸分子段上的三个碱基在平面内的单向排列不同。比如:平面内的AAG排列与AGA排列分别是该三核苷酸分子段在平面内的两个不同形象,虽然排列不同但DNA链上的三核苷酸分子段相同。这就是化学史上著名的二氯甲烷的异构体个数问题 [7](这一问题的争论导致了立体化学的诞生)。所以,43 密码表在具体应用中与立体化学的基本概念相矛盾。 (七)在Crick 43 遗传密码表理想应用情况下,给定生命体的DNA分子碱基序列完全按照43 遗传密码表指示下翻译为相应蛋白质的氨基酸顺序。这一过程,相当于按照“111元:脚蹬;211元:钢架;122元:车轮;112元:后座”的自行车零件价格表,购进材料,按照人为规定的自行车线性排列模型“钢架—车轮—车轮—后座—脚蹬”组装成一辆自行车,其结果是“1辆该模式的自行车,价值211,122,122,112,111元”,买零件只需要区区千元人民币,买一辆自行车要花上千万亿元人民币!——— 43 遗传密码表的这一应用方式是无法让人理解的!其错误的根源在于“用极不详尽的反应发生过程粗略地把43 密码表中的64年独立排列选任意多个后首尾相连起来,并进而错误地表达不确定碱基个数(但远大于192个碱基数)的DNA碱基序列的排列!所以,实际上,各个生命体的DNA分子与蛋白质分子间遗传密码结论和遗传密码表结论是完全错误的! 六、遗传学对43 密码表的中性态度 样例:成年人的血红蛋白分子由4条多肽链组成,其中两条各含141个氨基酸,称为a链,另外两条各含146个氨基酸,称为β链。β链第6位氨基酸的改变可以导致血红蛋白的功能发生改变,表现为镰刀型贫血症。科学家曾经用分子生物学手段检测到某一对夫妇的一对血红蛋白基因完全相同,编码β链第6位氨基酸的等位DNA碱基有一个是正常的GAG,另一个是异常的GTG。在这里我们可以用β/βS这样的符号表示这一对等位基因,其中β为正常基因,βS为异常基因。科学家又检测了这对夫妇所生的3个孩子,结果一个是β/βS,再一个是β/β,又一个是βS / βS。 以样例为,遗传学上更喜欢用组合数的方法研究遗传的规律性。本文简要论述以下几个方面: (一)遗传学基因分型的基本概念 “纯合基因”和“杂合基因”,是基因种类相同和不相同的专一描述,这一描述的数学特性是“注重元素是否相同,而不注重元素相互间的顺序”。这是数学上组合数知识的基本特征,不是排列数知识的特征。 (二)基因型杂合情况下,根据个体表现基因的情况,将之分为“显性基因”、“隐性基因”等情况,而不注重X1 X2 X3 X4…Xn个显性基因(或Y1, Y2, Y3…Yn个隐性基因)在杂合基因型中的排列顺序问题,所以,显性基因和隐性基因的概念也是组合数知识的运用,不是排列数知识的运用。 (三)从密码表的“遗传”表头和实验论证上“DNA~蛋白质”间“反应物~生成物”关系看,可以说“DNA~蛋白质”关系满足遗传学上讲的“父代~子代”关系。43 密码表在遗传学上的应用意味着子代(蛋白质)全部信息由父代(DNA分子)而来,而这一应用性结论远远违背传统遗传学上讲的父代基因优化组合传递给子代(基因图谱的本质)的选择性本质。 结 尾 基于43 遗传密码表从产生到运用发生的系列错误,同时根据碰撞理论等反应机制的内在需求,笔者建议化学家和数学家联手建立一个平面内物质分子关于其构成原子的排列数模型(用平面上的排列数语言描述物质分子的空间几何形象)和反应发生时生成物分子关于反应物分子的排列数模型(用反应物的排列数语言而不是组合数语言描述反应发生的机理),从而保证排列数方法在化学反应中的正确运用。 参考:1、 化学会第24届学术年会,2004年4月《新世纪的中国化学:机会与挑战》:04-0812 、 张文彦、支继军、张继光主编,科学技术文献出版社,1992年,《自然科学大事典》:548-5493 、北京农业大学 主编,农业出版社,1982年,《植物生》:464、 同济大学数学教研室 编,高等出版社,1998年,《线性代数》:635、 [英] B.F.C.克拉克 著,钟安环译,科学出版社,1982年,《遗传密码》:366、 [美] Lubert Stryer 著 唐有祺、张惠珠、吴相钰等译校,北京大学出版社, 《生物化学》:486-4887、 邢其毅、徐瑞秋等 著,高等教育出版社,2002年,《有机化学》:13
对第二个CUU应该跳读,实际应用中继续把第31个CUU读为亮氨酸。 (五)密码表应用性结论“高度重复序列和中度重复序列不编码蛋白质”与密码表引以确立的经典实验“PQRPQRPQR…PQR高度重复序列确立PQR→aa1,QRP→aa2 , RQP→aa3 , 三类密码子破译”完全相违背。这说明,Khorana合成的有确定顺序的核苷酸链用于破译密码子氨基酸意义的方法在具体应用中行不通。因而Khorana 的PQRPQRPQR…PQR法所确立的大量密码子的氨基酸意义是不准确的,是不可靠的。 (六) 若DNA链上某个三核苷酸分子段以分子模型形式独立存在,且在空间某域的相位不变,那么,观察者所站角度不同,该三核苷酸分子段上的三个碱基在平面内的单向排列不同。比如:平面内的AAG排列与AGA排列分别是该三核苷酸分子段在平面内的两个不同形象,虽然排列不同但DNA链上的三核苷酸分子段相同。这就是化学史上著名的二氯甲烷的异构体个数问题 [7](这一问题的争论导致了立体化学的诞生)。所以,43 密码表在具体应用中与立体化学的基本概念相矛盾。 (七)在Crick 43 遗传密码表理想应用情况下,给定生命体的DNA分子碱基序列完全按照43 遗传密码表指示下翻译为相应蛋白质的氨基酸顺序。这一过程,相当于按照“111元:脚蹬;211元:钢架;122元:车轮;112元:后座”的自行车零件价格表,购进材料,按照人为规定的自行车线性排列模型“钢架—车轮—车轮—后座—脚蹬”组装成一辆自行车,其结果是“1辆该模式的自行车,价值211,122,122,112,111元”,买零件只需要区区千元人民币,买一辆自行车要花上千万亿元人民币!——— 43 遗传密码表的这一应用方式是无法让人理解的!其错误的根源在于“用极不详尽的反应发生过程粗略地把43 密码表中的64年独立排列选任意多个后首尾相连起来,并进而错误地表达不确定碱基个数(但远大于192个碱基数)的DNA碱基序列的排列!所以,实际上,各个生命体的DNA分子与蛋白质分子间遗传密码结论和遗传密码表结论是完全错误的! 六、遗传学对43 密码表的中性态度 样例:成年人的血红蛋白分子由4条多肽链组成,其中两条各含141个氨基酸,称为a链,另外两条各含146个氨基酸,称为β链。β链第6位氨基酸的改变可以导致血红蛋白的功能发生改变,表现为镰刀型贫血症。科学家曾经用分子生物学手段检测到某一对夫妇的一对血红蛋白基因完全相同,编码β链第6位氨基酸的等位DNA碱基有一个是正常的GAG,另一个是异常的GTG。在这里我们可以用β/βS这样的符号表示这一对等位基因,其中β为正常基因,βS为异常基因。科学家又检测了这对夫妇所生的3个孩子,结果一个是β/βS,再一个是β/β,又一个是βS / βS。 以样例为,遗传学上更喜欢用组合数的方法研究遗传的规律性。本文简要论述以下几个方面: (一)遗传学基因分型的基本概念 “纯合基因”和“杂合基因”,是基因种类相同和不相同的专一描述,这一描述的数学特性是“注重元素是否相同,而不注重元素相互间的顺序”。这是数学上组合数知识的基本特征,不是排列数知识的特征。 (二)基因型杂合情况下,根据个体表现基因的情况,将之分为“显性基因”、“隐性基因”等情况,而不注重X1 X2 X3 X4…Xn个显性基因(或Y1, Y2, Y3…Yn个隐性基因)在杂合基因型中的排列顺序问题,所以,显性基因和隐性基因的概念也是组合数知识的运用,不是排列数知识的运用。 (三)从密码表的“遗传”表头和实验论证上“DNA~蛋白质”间“反应物~生成物”关系看,可以说“DNA~蛋白质”关系满足遗传学上讲的“父代~子代”关系。43 密码表在遗传学上的应用意味着子代(蛋白质)全部信息由父代(DNA分子)而来,而这一应用性结论远远违背传统遗传学上讲的父代基因优化组合传递给子代(基因图谱的本质)的选择性本质。 结 尾 基于43 遗传密码表从产生到运用发生的系列错误,同时根据碰撞理论等反应机制的内在需求,笔者建议化学家和数学家联手建立一个平面内物质分子关于其构成原子的排列数模型(用平面上的排列数语言描述物质分子的空间几何形象)和反应发生时生成物分子关于反应物分子的排列数模型(用反应物的排列数语言而不是组合数语言描述反应发生的机理),从而保证排列数方法在化学反应中的正确运用。 参考:1、 化学会第24届学术年会,2004年4月《新世纪的中国化学:机会与挑战》:04-0812 、 张文彦、支继军、张继光主编,科学技术文献出版社,1992年,《自然科学大事典》:548-5493 、北京农业大学 主编,农业出版社,1982年,《植物生》:464、 同济大学数学教研室 编,高等出版社,1998年,《线性代数》:635、 [英] B.F.C.克拉克 著,钟安环译,科学出版社,1982年,《遗传密码》:366、 [美] Lubert Stryer 著 唐有祺、张惠珠、吴相钰等译校,北京大学出版社, 《生物化学》:486-4887、 邢其毅、徐瑞秋等 著,高等教育出版社,2002年,《有机化学》:13
上一篇:特需病房护理特色探讨











